最近我们被客户要求撰写关于多重比较的研究报告,包括一些图形和统计输出。假设检验的基本原理是小概率原理,即我们认为小概率事件在一次试验中实际上不可能发生。
当同一研究问题下进行多次假设检验时,不再符合小概率原理所说的“一次试验”。如果在该研究问题下只要有检验是阳性的,就对该问题下阳性结论的话,对该问题的检验的犯一类错误的概率就会增大。
多重比较的问题
如果同一问题下进行n次检验,每次的检验水准为α(每次假阳性概率为α),则n次检验至少出现一次假阳性的概率会比α大。假设每次检验独立的条件下该概率可增加至
可下载资源
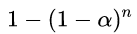
常见的多重比较情景包括:
1.什么是Familywise Error Rate(FWE or FWER)
定义:在一系列假设检验中,至少得出一次错误结论的概率。
换句话说,是造成至少一次Type I Error的概率。术语FWE来自测试系列,这是一系列数据测试的技术定义
2.估计FWE的公式
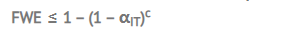
-
αIT:一个独立测试的显著水平
-
c : 对比次数
举个例子,对于一个10次试验的的序列,显著水平5%,FWE = ≤ 1 – (1 – .05)^10 = 0.401 ,这意味着Type I Error 发生的概率超过了40%,对于只有10次试验而言,是非常高的
3.控制FWE
单步执行,Bonferroni校正
单步程序对每个p值进行相同的调整。这保持了整个alpha水平保持在期望值(例如..05)。该方法被称为被称为Bonferroni校正。
1. 将alpha级别除以正在运行的测试的数量,并将该alpha级别应用于每个单独的测试。例如,如果您的整体alpha水平是.05,并且您正在运行5个测试,那么每个测试的alpha水平将是.05/5=.01。
2. 在每个测试中应用新的alpha级别来查找p值。在本例中,p值必须小于或等于0.01才具有统计意义。
Type I Error
Type I Error 就是错误地拒绝了一个真实的零假设Ho(null hypothesis)(应该接受的)
null hypothesis :普遍接受的假设
Ho是一个普遍接受的假设;它与备择假设相反。研究人员提出了另一种假设,他们认为这种假设解释了一种现象,然后努力拒绝零假设。
Type II Error
Il型错误(有时称为2型错误)是拒绝虚假零假设的失败。类型ll错误的概率用符号B表示。
- 多组间比较
- 多个主要指标
- 临床试验中期中分析
- 亚组分析
控制多重比较谬误(Familywise error rate):Bonferroni矫正
Bonferroni法得到的矫正P值=P×n
Bonferroni法非常简单,它的缺点在于非常保守(大概是各种方法中最保守的了),尤其当n很大时,经过Bonferroni法矫正后总的一类错误可能会远远小于既定α。
控制错误发现率:Benjamini & Hochberg法
简称BH法。首先将各P值从小到大排序,生成顺序数
排第k的矫正P值=P×n/k
另外要保证矫正后的各检验的P值大小顺序不发生变化。
怎么做检验
R内置了一些方法来调整一系列p值,以控制多重比较谬误(Familywise error rate)或控制错误发现率。
Holm、Hochberg、Hommel和Bonferroni方法控制了多重比较谬误(Familywise error rate)。这些方法试图限制错误发现的概率(I型错误,在没有实际效果时错误地拒绝无效假设),因此都是相对较保守的。
方法BH(Benjamini-Hochberg,与R中的FDR相同)和BY(Benjamini & Yekutieli)控制错误发现率,这些方法试图控制错误发现的期望比例。
请注意,这些方法只需要调整p值和要比较的p值的数量。这与Tukey或Dunnett等方法不同,Tukey和Dunnett也需要基础数据的变异性。Tukey和Dunnett被认为是多重比较谬误(Familywise error rate)方法。
要了解这些不同调整的保守程度,请参阅本文下面的两个图。
关于使用哪种p值调整度量没有明确的建议。一般来说,你应该选择一种你的研究领域熟悉的方法。此外,可能有一些逻辑允许你选择如何平衡犯I型错误和犯II型错误的概率。例如,在一项初步研究中,你可能希望保留尽可能多的显著值,来避免在未来的研究中排除潜在的显著因素。另一方面,在危及生命并且治疗费用昂贵的医学研究中,得出一种治疗方法优于另一种治疗方法的结论之前,你应该有很高的把握。
具有25个p值的多重比较示例
### --------------------------------------------------------------
### 多重比较示例
### --------------------------------------------------------------
Data = read.table(Input,header=TRUE)
按p值排序数据
Data = Data[order(Data$Raw.p),] 
检查数据是否按预期的方式排序

执行p值调整并添加到数据框
Data$Bonferroni =
p.adjust(Data$Raw.p,
method = "bonferroni")
Data$BH =
p.adjust(Data$Raw.p,
method = "BH")
Data$Holm =
p.adjust(Data$ Raw.p,
method = "holm")
Data$Hochberg =
p.adjust(Data$ Raw.p,
method = "hochberg")
Data$Hommel =
p.adjust(Data$ Raw.p,
method = "hommel")
Data$BY =
p.adjust(Data$ Raw.p,
method = "BY")
Data 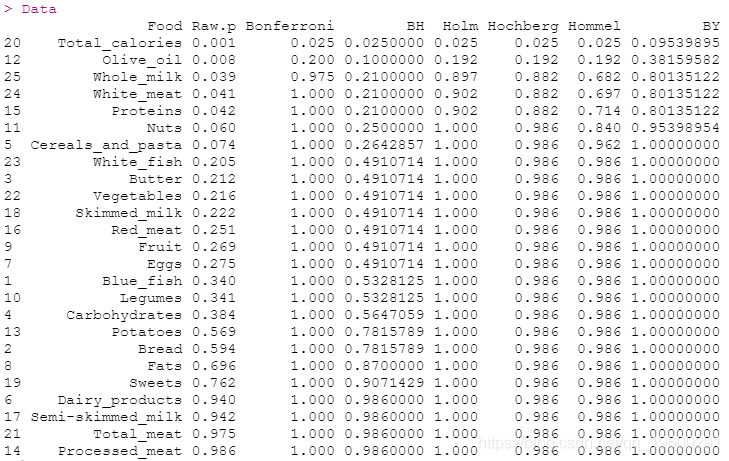
绘制图表
plot(X, Y,
xlab="原始的p值",
ylab="矫正后的P值"
lty=1,
lwd=2随时关注您喜欢的主题
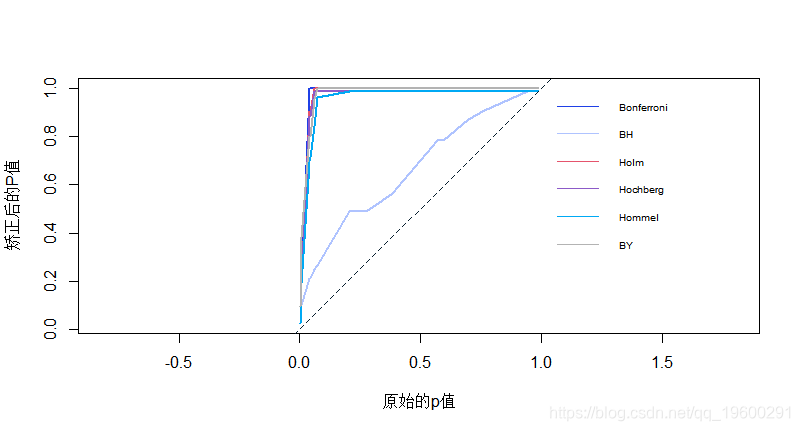
调整后的p值与原始的p值的图为一系列的25个p值。虚线表示一对一的线。
5个p值的多重比较示例
### --------------------------------------------------------------
### 多重比较示例,假设示例
### --------------------------------------------------------------
Data = read.table(Input,header=TRUE)执行p值调整并添加到数据帧
Data$Bonferroni =
p.adjust(Data$Raw.p,
method = "bonferroni")
Data$BH =
signif(p.adjust(Data$Raw.p,
method = "BH"),
4)
Data$Holm =
p.adjust(Data$ Raw.p,
method = "holm")
Data$Hochberg =
p.adjust(Data$ Raw.p,
method = "hochberg")
Data$Hommel =
p.adjust(Data$ Raw.p,
method = "hommel")
Data$BY =
signif(p.adjust(Data$ Raw.p,
method = "BY"),
4)
Data 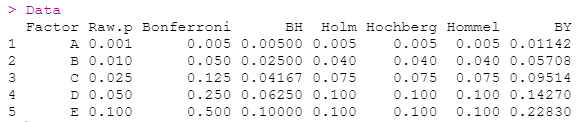
调整后的p值与原始p值在0到0.1之间的一系列5个p值的绘图。请注意,Holm和Hochberg的值与Hommel相同,因此被Hommel隐藏。虚线表示一对一的线。
绘制(图表)
plot(X, Y,
type="l", 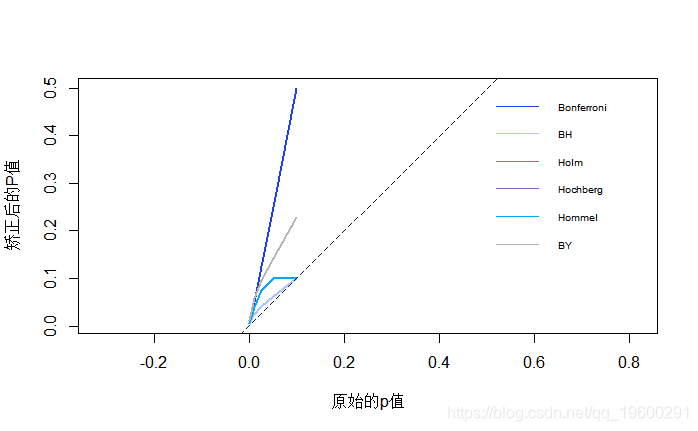



 【视频】因子分析简介及R语言应用实例:对地区经济研究分析重庆市经济指标
【视频】因子分析简介及R语言应用实例:对地区经济研究分析重庆市经济指标 R语言宏观经济学:IS-LM曲线可视化货币市场均衡
R语言宏观经济学:IS-LM曲线可视化货币市场均衡 R语言代做编程辅导和解答GLM Coursework
R语言代做编程辅导和解答GLM Coursework R语言分析上海空气质量数据:kmean聚类、层次聚类、时间序列分析:arima模型、指数平滑法
R语言分析上海空气质量数据:kmean聚类、层次聚类、时间序列分析:arima模型、指数平滑法


