Glmnet是一个通过惩罚最大似然关系拟合广义线性模型的软件包。
正则化路径是针对正则化参数λ的值网格处的lasso或Elastic Net(弹性网络)惩罚值计算的。
该算法非常快,并且可以利用输入矩阵中的稀疏性 x。它适合线性,逻辑和多项式,泊松和Cox回归模型。可以从拟合模型中做出各种预测。
视频
Lasso回归、岭回归等正则化回归数学原理及R语言实例
它也可以拟合多元线性回归。
glmnet 解决以下问题

在覆盖整个范围的λ值网格上。这里l(y,η)是观察i的负对数似然贡献;例如对于高斯分布是![]() 。 弹性网络惩罚由α控制,LASSO(α= 1,默认),Ridge(α= 0)。调整参数λ控制惩罚的总强度。
。 弹性网络惩罚由α控制,LASSO(α= 1,默认),Ridge(α= 0)。调整参数λ控制惩罚的总强度。
一、用途
正则化Regularization主要用来防止overfitting。
下面举例会发生的一些模型评估情况:
-
比如在KNN(K-Nearest Neighbors)中,单纯用knn.score作模型评估会忽视样本不平衡情况,导致评估正确率不准确。用代码knn.score得到是分子除以分母的形式:
。
这种测评model方法并不完美。因为有可能发生一种情况:不平衡样本比例,导致错评model。比如判断垃圾邮件分类,一共有100个测试数据集,只有5个是垃圾邮件。正确率有95%,即使无法判断垃圾邮件(预测有100%的正确率)与实际正确率也相差不大。可以采用confusion matrix 方法。(PS: 下一篇,会写各种评估模型性能的方法。)
2. 比如在线性回归Linner Regression 中,可以用代码.score得到,是R squared 衡量X与y相关性,它评价你选择的线性模型合适与否。如果R squared偏小,说明特征X的weight比重较小,还有其他的特征没有考虑进去。另外也可以采用均方根误差(RMSE)作为评价回归模型的另一个常用指标。
3. 对于评价模型首先我们会检验Bias-Variance-Tradeoff。
(1)如果模型Bias过高,说明模型的预测值与偏差值较大,模型欠拟合,应该选择更好的模型。为了充分学习训练集数据,解决欠拟合方法有:设置k-fold cross vaildation (交叉验证方法)。把数据集分成k份测试集。每次用剩下(k-1)份作为训练集,测试在1份测试集上。共进行K次验证。例如Linear regression中衡量cross_val_score是R squared。
(2)如果模型Variance过高,虽然对训练集高度学习,但太关注个体变动比如学到了Noise,模型过拟合。考虑过拟合原因:
-
特征选取过多 ,导致参数过多。
-
部分特征的权重或者它的系数过大(the coefficient or the weight of features too large)
正则化Regularization的产生是为了解决改善上述过拟合源头。
二、L1, L2 及ElasticNet弹性网络回归概念
正则化内有两步,分别是L1: Lasso;L2: Ridge。
概念:
-
L1称为Lasso,L1 Lasso回归是在损失函数基础上加入了绝对值和的正则化方法。
用途:主要选择出重要特征。
特征:使weight 趋向0,在特征选择上特别重要!
公式:
2. L2称为Ridge, L2 Ridge回归是在损失函数基础上加入了平方和的正则化方法。
用途:主要惩罚系数过大的特征。
特征: (正则化参数)越大,限制过拟合越强。
公式:
3. ElasticNet回归则是融合了两种正则方法,是Lasso回归与Ridge回归的组合。
公式:
注意点:
-
在一般步骤上,先L2 Ridge正则化方法,后L1 Lasso正则化方法。为什么呢?
存在这样一种情况,如果我们用来拟合的自变量过多(或者说特征变量过多),而且特征变量之前存在很高的相关关系,比如上面这种情况:以上两个函数都可以很好的拟合数据,但右边的函数显然有过拟合的嫌疑,为了避免这种情况,有两种方法:
方法一:舍掉x^3和x^4这两个变量(舍弃无用的特征变量)。这可以人工选择,也可以利用算法来做。但舍弃无用的特征变量会舍弃一部分数据,一部分数据可能是有意义的。所以推荐方法二。
方法二:减小θ3和θ4的值(即正则化,保留所有特征变量,但减少变量参数的值)。
结论:先采用Ridge回归,后采用Lasso回归是因为为了保护数据的完整性,先保留所有特征变量,但减少变量参数的权重值。而实际情况中,我们很难判断哪些特征变量需要正则化,所以一般情况下,我们是对所有的参数都正则化处理:
2. 其中 是正则项,
为正则参数, 不能设置过大。需要注意的是,i是从1开始的,这意味着函数的常数项(
)并没有被正则化。所以正则化
不能设的太大,否则会导致除了常数项外,所有的参数值都很小,因变量近似等于常数项,出现欠拟合现象。
三、由来原因
1. 从期望风险、经验风险与结构风险之间关系的角度
在训练集上做损失函数是经验风险,然而经验风险最小化无法保证未知数据集上的损失函数最小,同时理想的期望风险中的联合分布不可求。所以折中通过结构风险,使模型最优化。
正则化是结构风险中处理手段的一种。
无论经验风险、结构风险、期望风险,它们的基础都是损失函数(Loss Function)。
A. 经验风险
经验风险就是最普通的损失函数,对训练集内所有样本点求损失函数的平均和。
从单个样本点Loss function入手,它是针对单个具体的样本而言的,表示的是模型预测的值与样本真实值之间的差距。比如对于某个样本 ,其真实的值为
,而我们的模型选择决策函数为
。那么通过模型预测的值为
;损失函数就是用来表示
与
之间的差距的,我们用函数
来衡量。我们希望的是这个
函数最小化。理想的情况是模型决策函数预测值
刚好等于该样本的真值
。
通过损失函数我们能知道模型决策函数f(X)对于单个样本点的预测能力。损失函数 越小,说明模型对于该样本预测越准确。如果想知道模型f(X)对训练样本中所有的样本的预测能力只需所有的样本点都求一次损失函数然后进行累加就好了。
经验风险越小说明模型 对训练集的拟合程度越好。
但是对于未知的样本效果怎么样呢?我们知道未知的样本数据( )的数量是不容易确定的,所以就没有办法用所有样本损失函数的平均值的最小化这个方法。
B. 期望风险
期望风险是一种理想化状态,是对全体数据的模型预测。
如果想要评估全部数据(样本数据+未知数据),理想化的角度想到概率论中期望Expectation。即假设X和Y服从联合分布P(X,Y).那么期望风险就可以表示为:
(假设X和Y的所有数据服从联合分布)
为什么要引入损失函数的期望呢?人们希望模型能够刻画在全体样本上的预测能力!但是联合概率 往往是无法得到的,因为无法得到全体样本数据包括未知数据,这一过程必须知道输入为
的概率
, 及在输入为
的情况下输出是的概率
,才能知道联合概率
。但是给出的样本往往不能反映真实的分布情况,期望风险不能直接计算,也就没法做期望风险最小化。
但是根据弱大数定律,当样本N无限大时,可用经验风险作为期望风险的估计,也就是局部估计整体。那么我们常说的风险最小化其实就指的是经验风险最小化!
C. 结构风险
比较经验风险和期望风险:
-
经验风险是建立在训练集上所有点的损失最少,它是是建立在局部;而期望风险是建立在全部集上所有点的损失最少,它是建立在全局。
2. 经验风险是现实的、可求的;期望风险是不现实的、不可求的。
产生问题:如果只考虑经验风险,会产生过拟合情况。经验风险越小,说明样本拟合程度越好(高) high variance。 模型f(x)对训练集中所有的样本点都有最好的预测能力,但是对于非训练集中的样本数据,模型的预测能力非常不好。
这种情况引出结构风险,在经验风险基础上添加一个正则项,表达式是:
其中 正则化系数是一个大于0的系数。
表示的是是模型的复杂度(惩罚项)。结构风险可以这么理解:
经验风险越小,模型决策函数越复杂,其包含的参数越多,当经验风险函数小到一定程度就出现了过拟合现象。也可以理解为模型决策函数的复杂程度是过拟合的必要条件,那么我们要想防止过拟合现象的方式,就要破坏这个必要条件,即降低决策函数的复杂度。也即,让惩罚项 最小化,现在出现两个需要最小化的函数了。我们需要同时保证经验风险函数和模型决策函数的复杂度都达到最小化,一个简单的办法把两个式子融合成一个式子得到结构风险函数然后对这个结构风险函数进行最小化。
众所周知,岭惩罚使相关预测因子的系数彼此缩小,而套索倾向于选择其中一个而丢弃其他预测因子。弹性网络则将这两者混合在一起。
glmnet 算法使用循环坐标下降法,该方法在每个参数固定不变的情况下连续优化目标函数,并反复循环直到收敛,我们的算法可以非常快速地计算求解路径。
代码可以处理稀疏的输入矩阵格式,以及系数的范围约束,还包括用于预测和绘图的方法,以及执行K折交叉验证的功能。
快速开始
首先,我们加载 glmnet 包:
library(glmnet)包中使用的默认模型是高斯线性模型或“最小二乘”。我们加载一组预先创建的数据以进行说明。用户可以加载自己的数据,也可以使用工作空间中保存的数据。
该命令 从此保存的R数据中加载输入矩阵 x 和因向量 y。
我们拟合模型 glmnet。
fit = glmnet(x, y)可以通过执行plot 函数来可视化系数 :
plot(fit)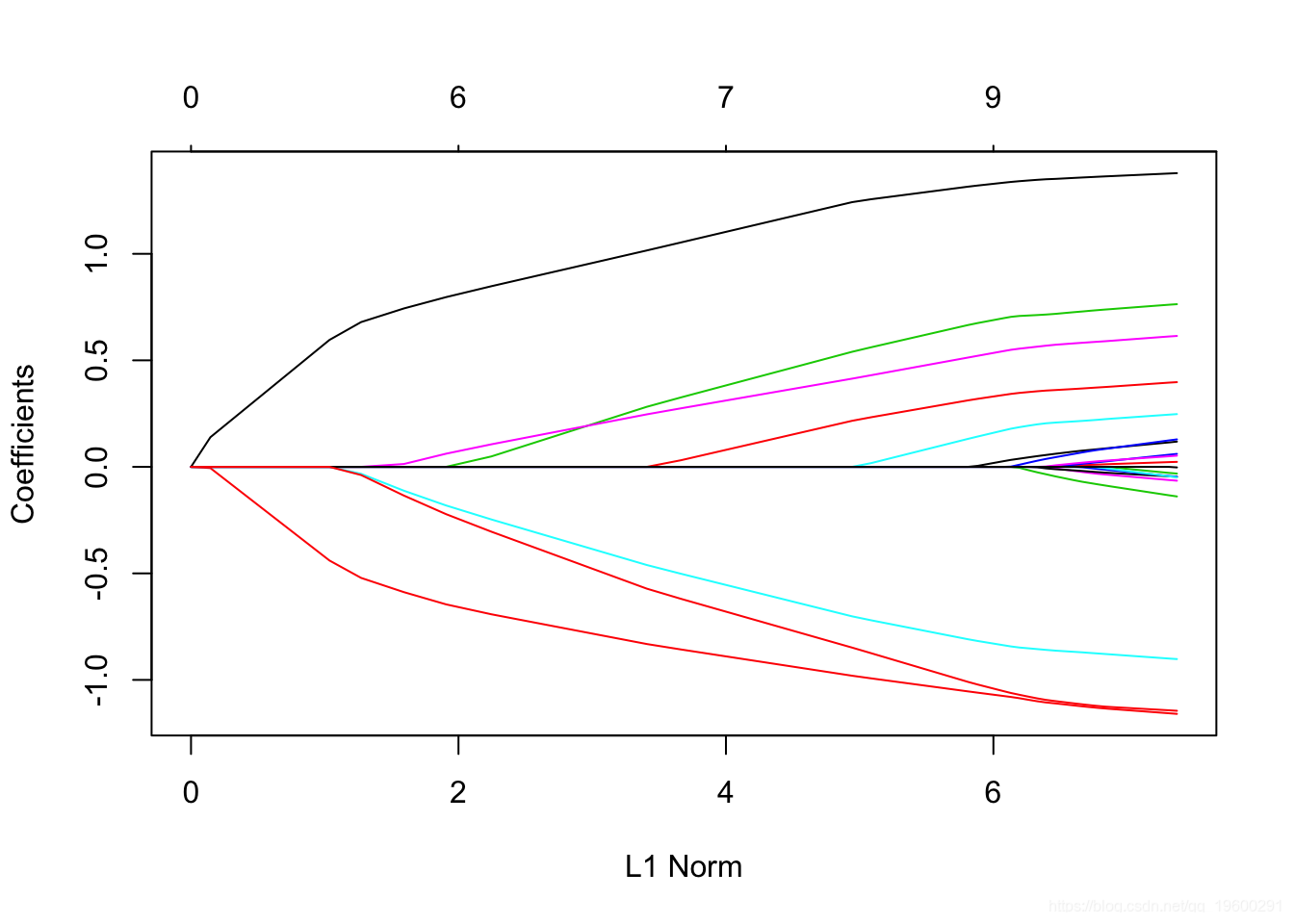
每条曲线对应一个变量。它显示了当λ变化时,其系数相对于整个系数向量的ℓ1范数的路径。上方的轴表示当前λ处非零系数的数量,这是套索的有效自由度(df)。用户可能还希望对曲线进行注释。这可以通过label = TRUE 在plot命令中进行设置来完成 。
glmnet 如果我们只是输入对象名称或使用print 函数,则会显示每个步骤的路径 摘要 :
print(fit)##
## Call: glmnet(x = x, y = y)
##
## Df %Dev Lambda
## [1,] 0 0.0000 1.63000
## [2,] 2 0.0553 1.49000
## [3,] 2 0.1460 1.35000
## [4,] 2 0.2210 1.23000
## [5,] 2 0.2840 1.12000
## [6,] 2 0.3350 1.02000
## [7,] 4 0.3900 0.93300
## [8,] 5 0.4560 0.85000
## [9,] 5 0.5150 0.77500
## [10,] 6 0.5740 0.70600
## [11,] 6 0.6260 0.64300
## [12,] 6 0.6690 0.58600
## [13,] 6 0.7050 0.53400
## [14,] 6 0.7340 0.48700
## [15,] 7 0.7620 0.44300
## [16,] 7 0.7860 0.40400
## [17,] 7 0.8050 0.36800
## [18,] 7 0.8220 0.33500
## [19,] 7 0.8350 0.30600
## [20,] 7 0.8460 0.27800它从左到右显示了非零系数的数量(Df),解释的(零)偏差百分比(%dev)和λ(Lambda)的值。
我们可以在序列范围内获得一个或多个λ处的实际系数:
coef(fit,s=0.1)## 21 x 1 sparse Matrix of class "dgCMatrix"
## 1
## (Intercept) 0.150928
## V1 1.320597
## V2 .
## V3 0.675110
## V4 .
## V5 -0.817412
## V6 0.521437
## V7 0.004829
## V8 0.319416
## V9 .
## V10 .
## V11 0.142499
## V12 .
## V13 .
## V14 -1.059979
## V15 .
## V16 .
## V17 .
## V18 .
## V19 .
## V20 -1.021874还可以使用新的输入数据在特定的λ处进行预测:
predict(fit,newx=nx,s=c(0.1,0.05))## 1 2
## [1,] 4.4641 4.7001
## [2,] 1.7509 1.8513
## [3,] 4.5207 4.6512
## [4,] -0.6184 -0.6764
## [5,] 1.7302 1.8451
## [6,] 0.3565 0.3512
## [7,] 0.2881 0.2662
## [8,] 2.7776 2.8209
## [9,] -3.7016 -3.7773
## [10,] 1.1546 1.1067该函数 glmnet 返回一系列模型供用户选择。交叉验证可能是该任务最简单,使用最广泛的方法。
cv.glmnet 是交叉验证的主要函数。
cv.glmnet 返回一个 cv.glmnet 对象,此处为“ cvfit”,其中包含交叉验证拟合的所有成分的列表。
我们可以绘制对象。
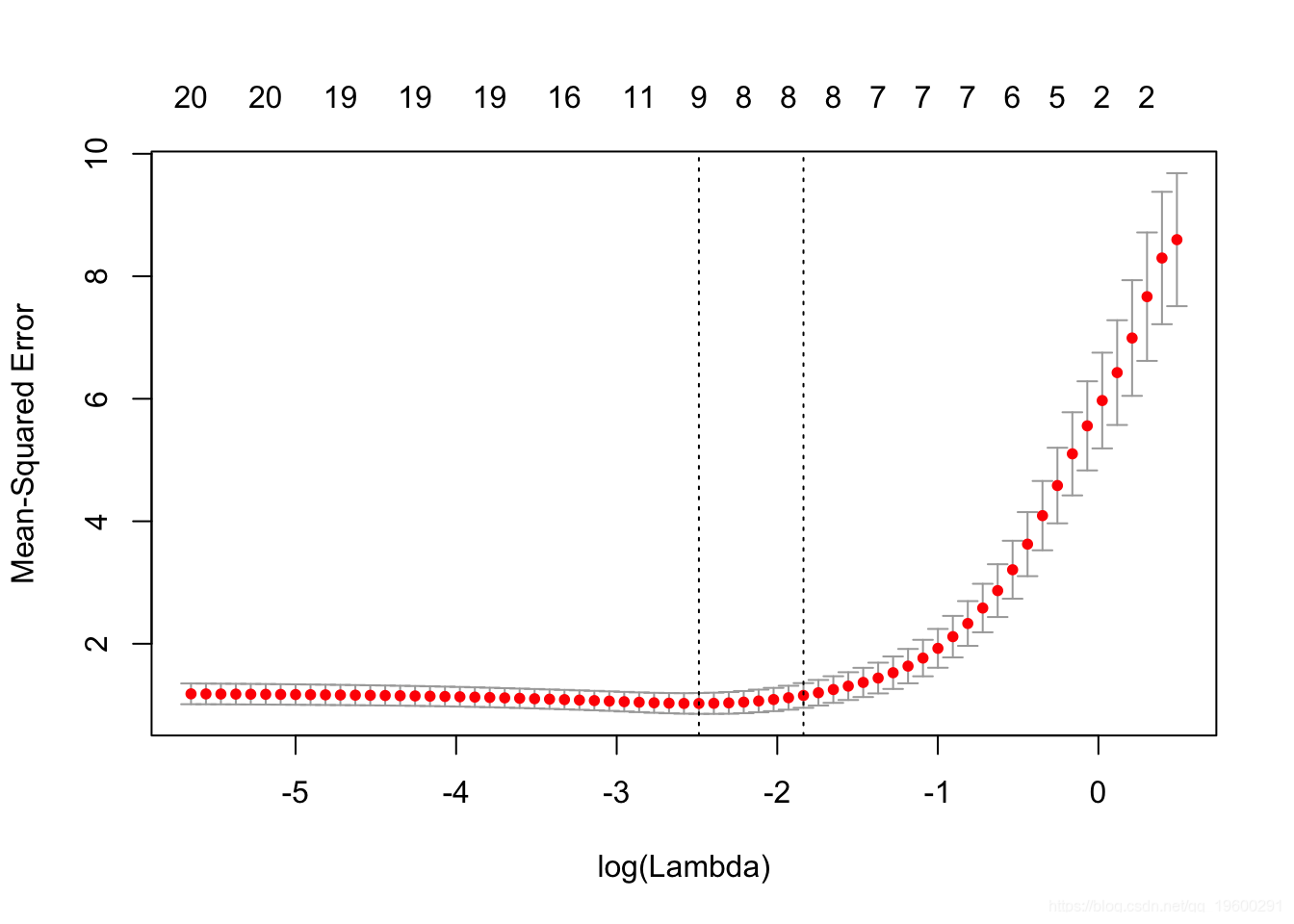
它包括交叉验证曲线(红色虚线)和沿λ序列的上下标准偏差曲线(误差线)。垂直虚线表示两个选定的λ。
我们可以查看所选的λ和相应的系数。例如,
cvfit$lambda.min## [1] 0.08307lambda.min 是给出最小平均交叉验证误差的λ值。保存的另一个λ是 lambda.1se,它给出了的模型,使得误差在最小值的一个标准误差以内。我们只需要更换 lambda.min 到lambda.1se 以上。
coef(cvfit, s = "lambda.min")## 21 x 1 sparse Matrix of class "dgCMatrix"
## 1
## (Intercept) 0.14936
## V1 1.32975
## V2 .
## V3 0.69096
## V4 .
## V5 -0.83123
## V6 0.53670
## V7 0.02005
## V8 0.33194
## V9 .
## V10 .
## V11 0.16239
## V12 .
## V13 .
## V14 -1.07081
## V15 .
## V16 .
## V17 .
## V18 .
## V19 .
## V20 -1.04341注意,系数以稀疏矩阵格式表示。原因是沿着正则化路径的解通常是稀疏的,因此使用稀疏格式在时间和空间上更为有效。
可以根据拟合的cv.glmnet 对象进行预测 。让我们看一个示例。
## 1## [1,] -1.3647## [2,] 2.5686## [3,] 0.5706## [4,] 1.9682## [5,] 1.4964newx 与新的输入矩阵 s相同,如前所述,是预测的λ值。
线性回归
这里的线性回归是指两个模型系列。一个是 gaussian正态分布,另一个是 mgaussian多元正态分布。
正态分布
假设我们有观测值xi∈Rp并且yi∈R,i = 1,…,N。目标函数是

其中λ≥0是复杂度参数,0≤α≤1在岭回归(α=0)和套索LASSO(α=1)之间。
应用坐标下降法解决该问题。具体地说,通过计算βj=β〜j处的梯度和简单的演算,更新为

其中![]() 。
。
当x 变量标准化为具有单位方差(默认值)时,以上公式适用 。
glmnet 提供各种选项供用户自定义。我们在这里介绍一些常用的选项,它们可以在glmnet 函数中指定 。
alpha表示弹性网混合参数α,范围α∈[0,1]。α=1是套索(默认),α=0是Ridge。weights用于观察权重。每个观察值的默认值为1。nlambda是序列中λ值的数量。默认值为100。lambda可以提供,但通常不提供,程序会构建一个序列。自动生成时,λ序列由lambda.max和 确定lambda.min.ratio。standardize是x在拟合模型序列之前进行变量标准化的逻辑标志 。
例如,我们设置α=0.2,并对后半部分的观测值赋予两倍的权重。为了避免在此处显示太长时间,我们将其设置 nlambda 为20。但是,实际上,建议将λ的数量设置为100(默认值)或更多。
然后我们可以输出glmnet 对象。
print(fit)##
## Call: glmnet(x = x, y = y, weights = c(rep(1, 50), rep(2, 50)), alpha = 0.2, nlambda = 20)
##
## Df %Dev Lambda
## [1,] 0 0.000 7.94000
## [2,] 4 0.179 4.89000
## [3,] 7 0.444 3.01000
## [4,] 7 0.657 1.85000
## [5,] 8 0.785 1.14000
## [6,] 9 0.854 0.70300
## [7,] 10 0.887 0.43300
## [8,] 11 0.902 0.26700
## [9,] 14 0.910 0.16400
## [10,] 17 0.914 0.10100
## [11,] 17 0.915 0.06230
## [12,] 17 0.916 0.03840
## [13,] 19 0.916 0.02360
## [14,] 20 0.916 0.01460
## [15,] 20 0.916 0.00896
## [16,] 20 0.916 0.00552
## [17,] 20 0.916 0.00340随时关注您喜欢的主题
这将显示生成对象的调用 fit 以及带有列Df (非零系数的数量), %dev (解释的偏差百分比)和Lambda (对应的λ值) 的三列矩阵 。
我们可以绘制拟合的对象。
让我们针对log-lambda值标记每个曲线来绘制“拟合”。
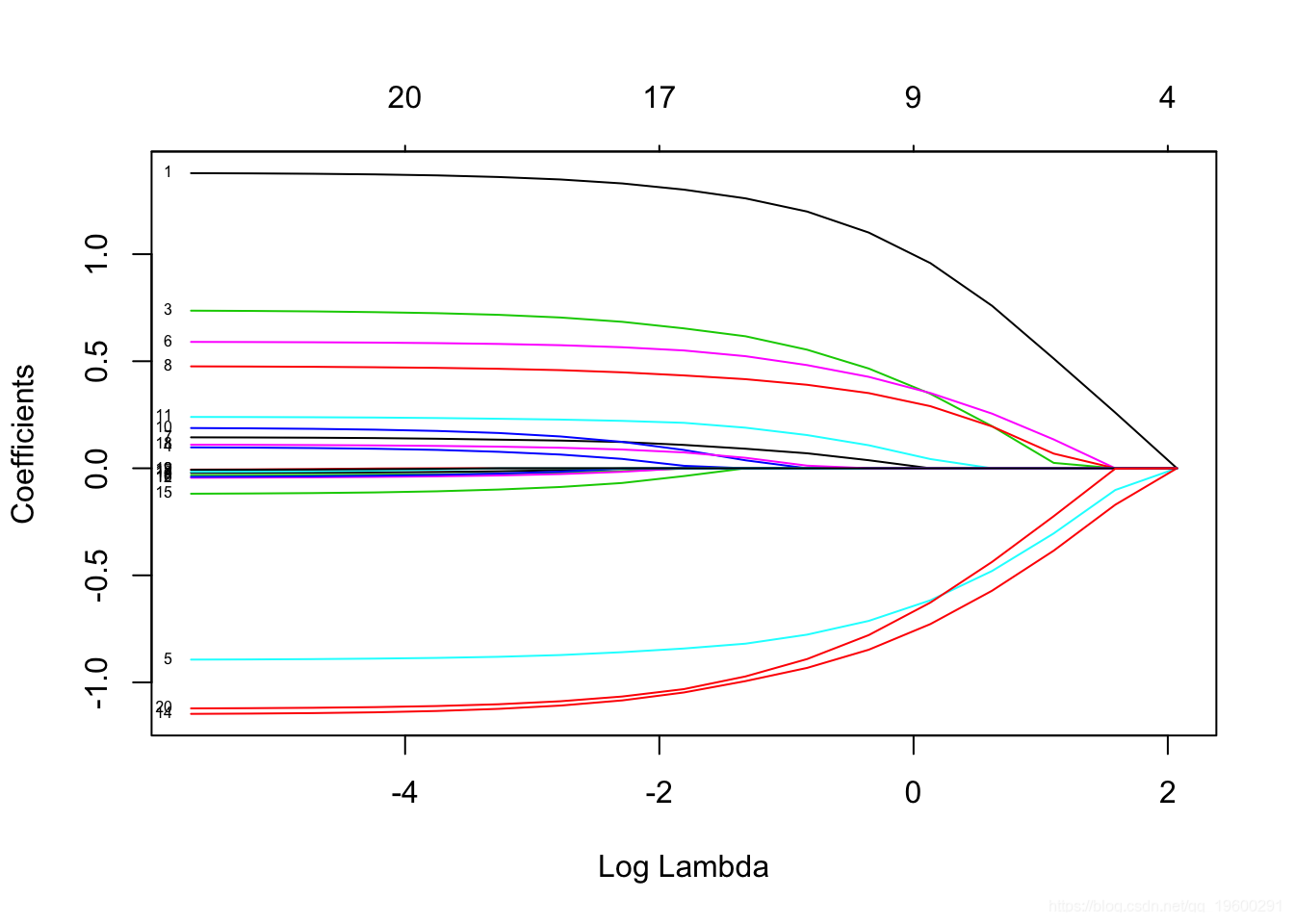
这是训练数据中的偏差百分比。我们在这里看到的是,在路径末端时,该值变化不大,但是系数有点“膨胀”。这使我们可以将注意力集中在重要的拟合部分上。
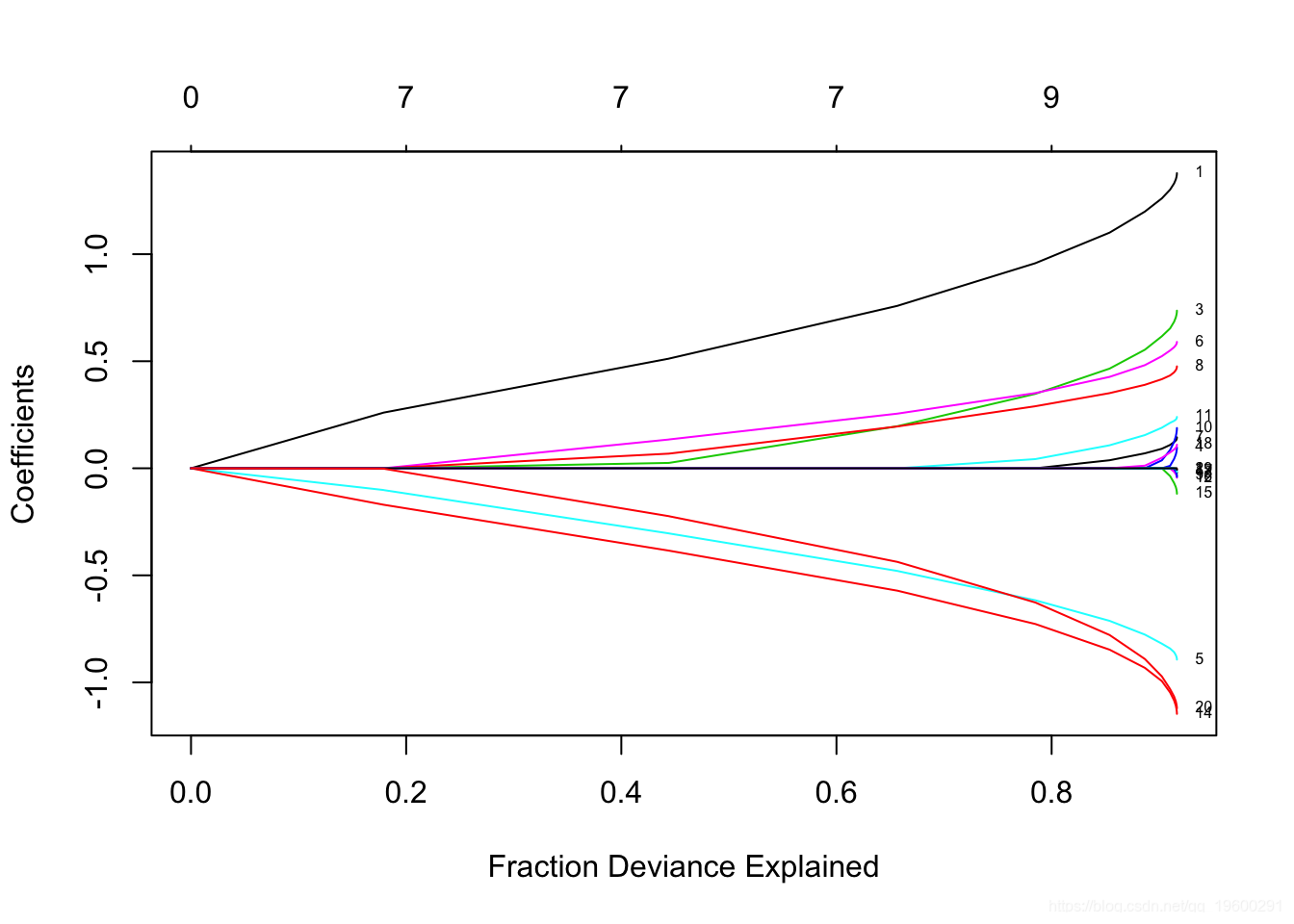
我们可以提取系数并在某些特定值的情况下进行预测。两种常用的选项是:
s指定进行提取的λ值。exact指示是否需要系数的精确值。
一个简单的例子是:
## 21 x 2 sparse Matrix of class "dgCMatrix"
## 1 1
## (Intercept) 0.19657 0.199099
## V1 1.17496 1.174650
## V2 . .
## V3 0.52934 0.531935
## V4 . .
## V5 -0.76126 -0.760959
## V6 0.46627 0.468209
## V7 0.06148 0.061927
## V8 0.38049 0.380301
## V9 . .
## V10 . .
## V11 0.14214 0.143261
## V12 . .
## V13 . .
## V14 -0.91090 -0.911207
## V15 . .
## V16 . .
## V17 . .
## V18 . 0.009197
## V19 . .
## V20 -0.86099 -0.863117左列是,exact = TRUE 右列是 FALSE。从上面我们可以看到,0.01不在序列中,因此尽管没有太大差异,但还是有一些差异。如果没有特殊要求,则线性插补就足够了。
用户可以根据拟合的对象进行预测。除中的选项外 coef,主要参数是 newx的新值矩阵 x。type 选项允许用户选择预测类型:*“链接”给出拟合值
- 因变量与正态分布的“链接”相同。
- “系数”计算值为的系数
s
例如,
## 1
## [1,] -0.9803
## [2,] 2.2992
## [3,] 0.6011
## [4,] 2.3573
## [5,] 1.7520给出在λ=0.05时前5个观测值的拟合值。如果提供的多个值, s 则会生成预测矩阵。
用户可以自定义K折交叉验证。除所有 glmnet 参数外, cv.glmnet 还有特殊的参数,包括 nfolds (次数), foldid (用户提供的次数), type.measure(用于交叉验证的损失):*“ deviance”或“ mse”
- “ mae”使用平均绝对误差
举个例子,
cvfit = cv.glmnet(x, y, type.measure = "mse", nfolds = 20)根据均方误差标准进行20折交叉验证。
并行计算也受 cv.glmnet。为我们在这里给出一个简单的比较示例。
system.time(cv.glmnet(X, Y))## user system elapsed
## 3.591 0.103 3.724system.time(cv.glmnet(X, Y, parallel = TRUE))## user system elapsed
## 4.318 0.391 2.700从上面的建议可以看出,并行计算可以大大加快计算过程。
- “ lambda.min”:达到最小MSE的λ。
cvfit$lambda.min## [1] 0.08307## 21 x 1 sparse Matrix of class "dgCMatrix"
## 1
## (Intercept) 0.14936
## V1 1.32975
## V2 .
## V3 0.69096
## V4 .
## V5 -0.83123
## V6 0.53670
## V7 0.02005
## V8 0.33194
## V9 .
## V10 .
## V11 0.16239
## V12 .
## V13 .
## V14 -1.07081
## V15 .
## V16 .
## V17 .
## V18 .
## V19 .
## V20 -1.04341在这里,我们使用相同的k折,为α选择一个值。
将它们全部放置在同一绘图上:
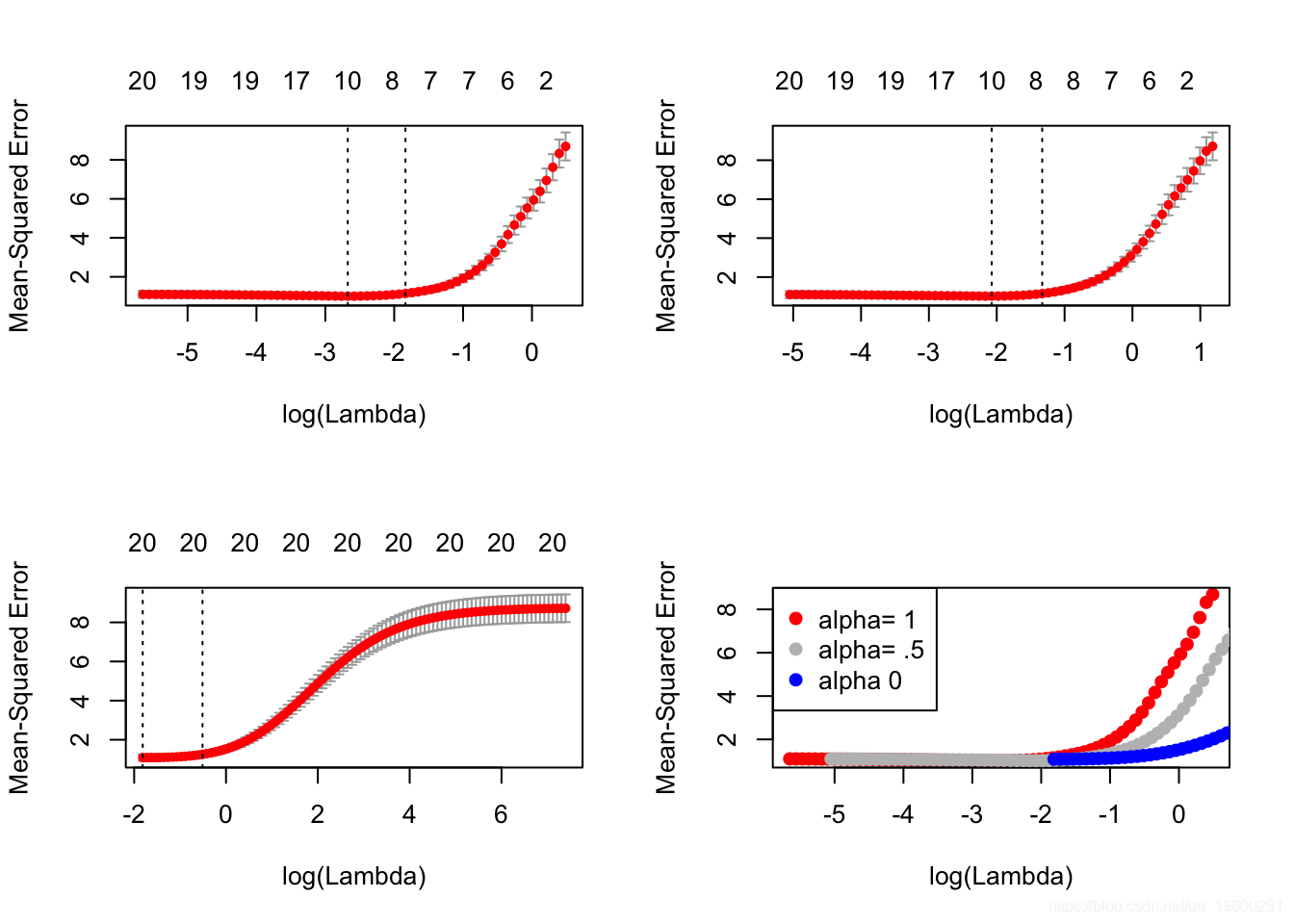
我们看到lasso(alpha=1)在这里表现最好。
系数上下限
假设我们要拟合我们的模型,但将系数限制为大于-0.7且小于0.5。这可以通过upper.limits 和 lower.limits 参数实现 :
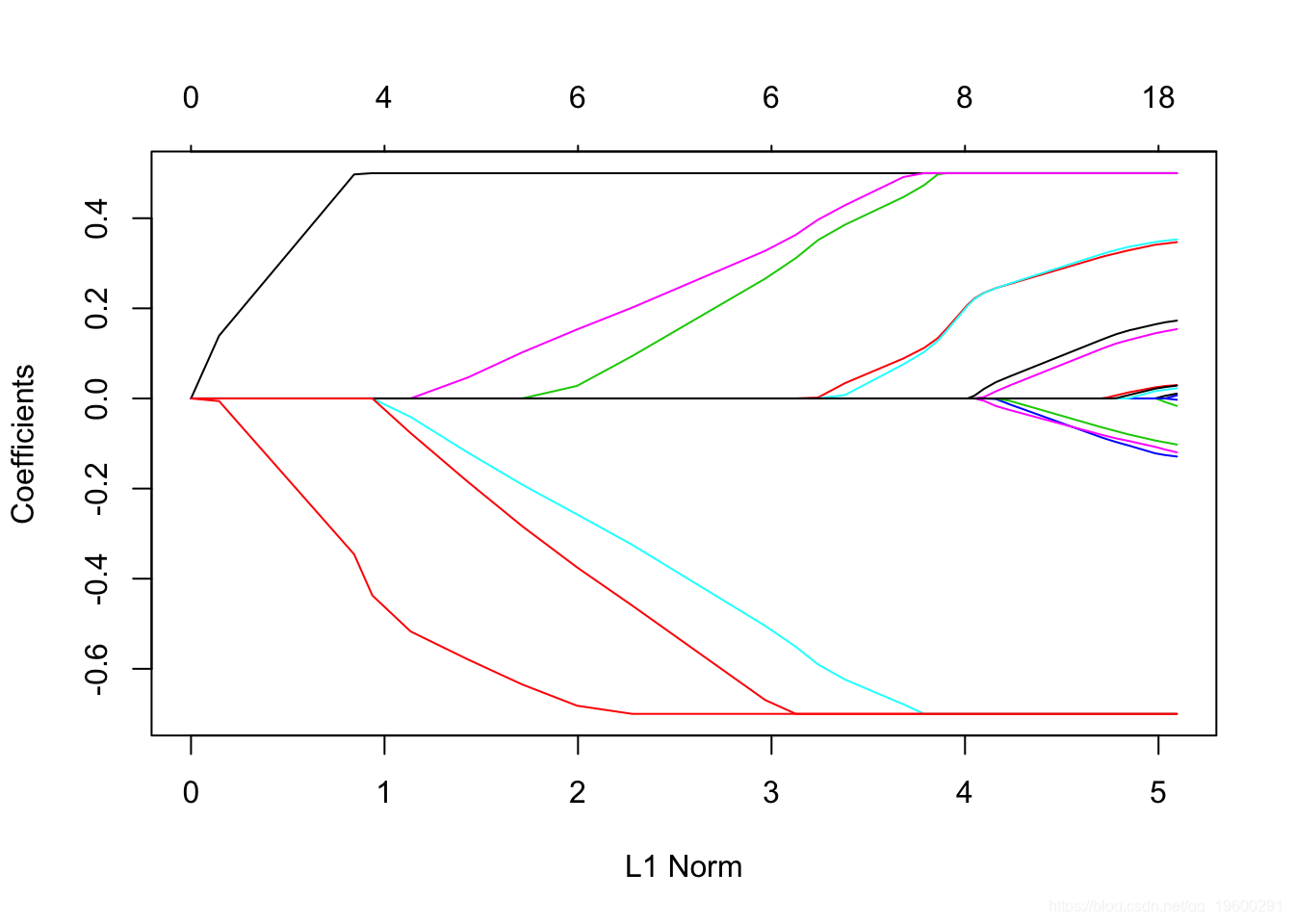
通常,我们希望系数为正,因此我们只能lower.limit 将其设置 为0。
惩罚因素
此参数允许用户将单独的惩罚因子应用于每个系数。每个参数的默认值为1,但可以指定其他值。特别是,任何penalty.factor 等于零的变量 都不会受到惩罚

在许多情况下,某些变量可能是重要,我们希望一直保留它们,这可以通过将相应的惩罚因子设置为0来实现:
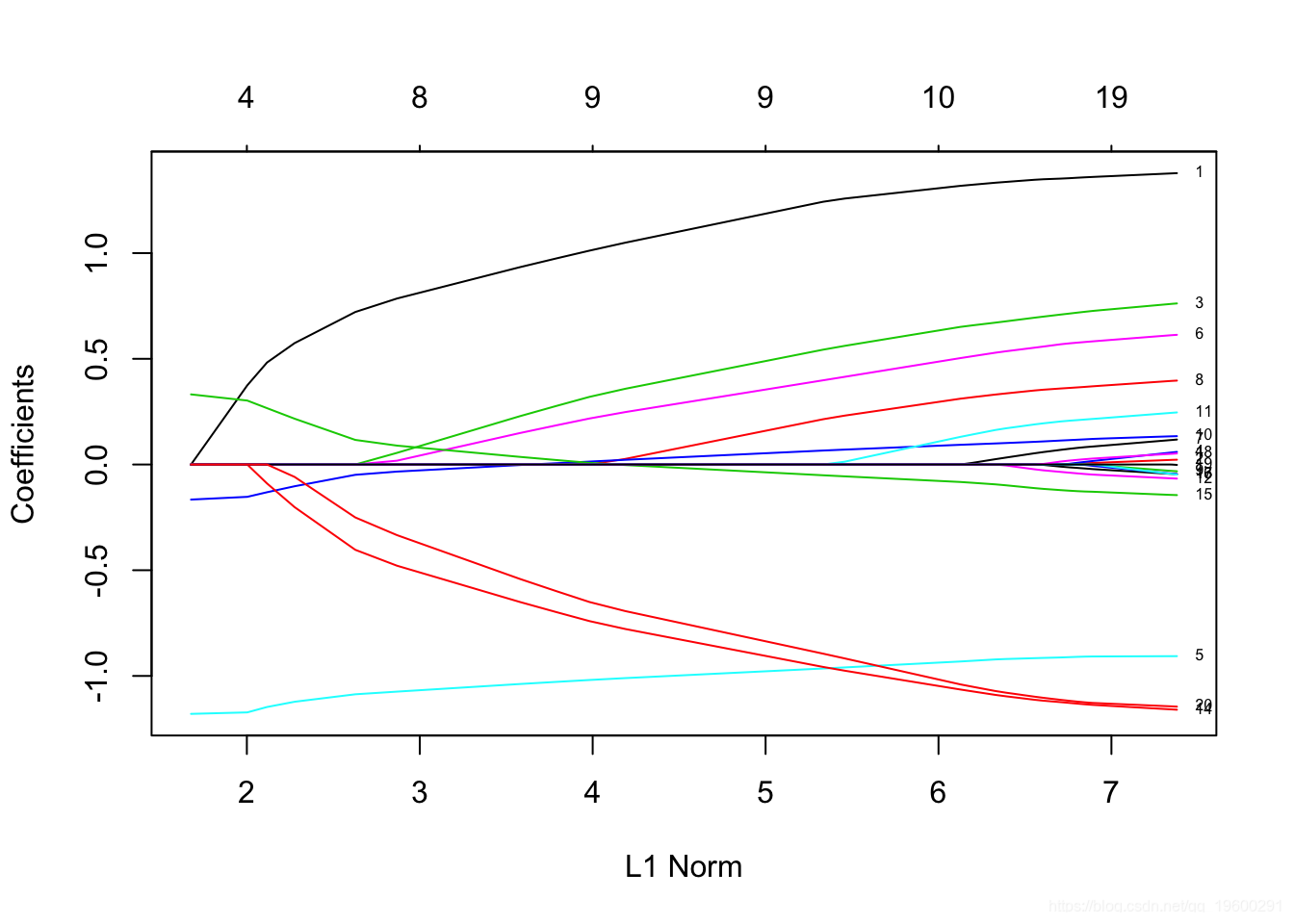
我们从标签中看到惩罚因子为0的三个变量始终保留在模型中,而其他变量遵循典型的正则化路径并最终缩小为0。
自定义图
有时,尤其是在变量数量很少的情况下,我们想在图上添加变量标签。
我们首先生成带有10个变量的一些数据,然后,我们拟合glmnet模型,并绘制标准图。

我们希望用变量名标记曲线。在路径的末尾放置系数的位置。

多元正态
使用family = "mgaussian" option 获得多元正态分布glmnet。
显然,顾名思义,y不是向量,而是矩阵。结果,每个λ值的系数也是一个矩阵。
在这里,我们解决以下问题:

这里,βj是p×K系数矩阵β的第j行,对于单个预测变量xj,我们用每个系数K向量βj的组套索罚分代替每个单一系数的绝对罚分。
我们使用预先生成的一组数据进行说明。
我们拟合数据,并返回对象“ mfit”。
mfit = glmnet(x, y, family = "mgaussian")如果为 standardize.response = TRUE,则将因变量标准化。
为了可视化系数,我们使用 plot 函数。
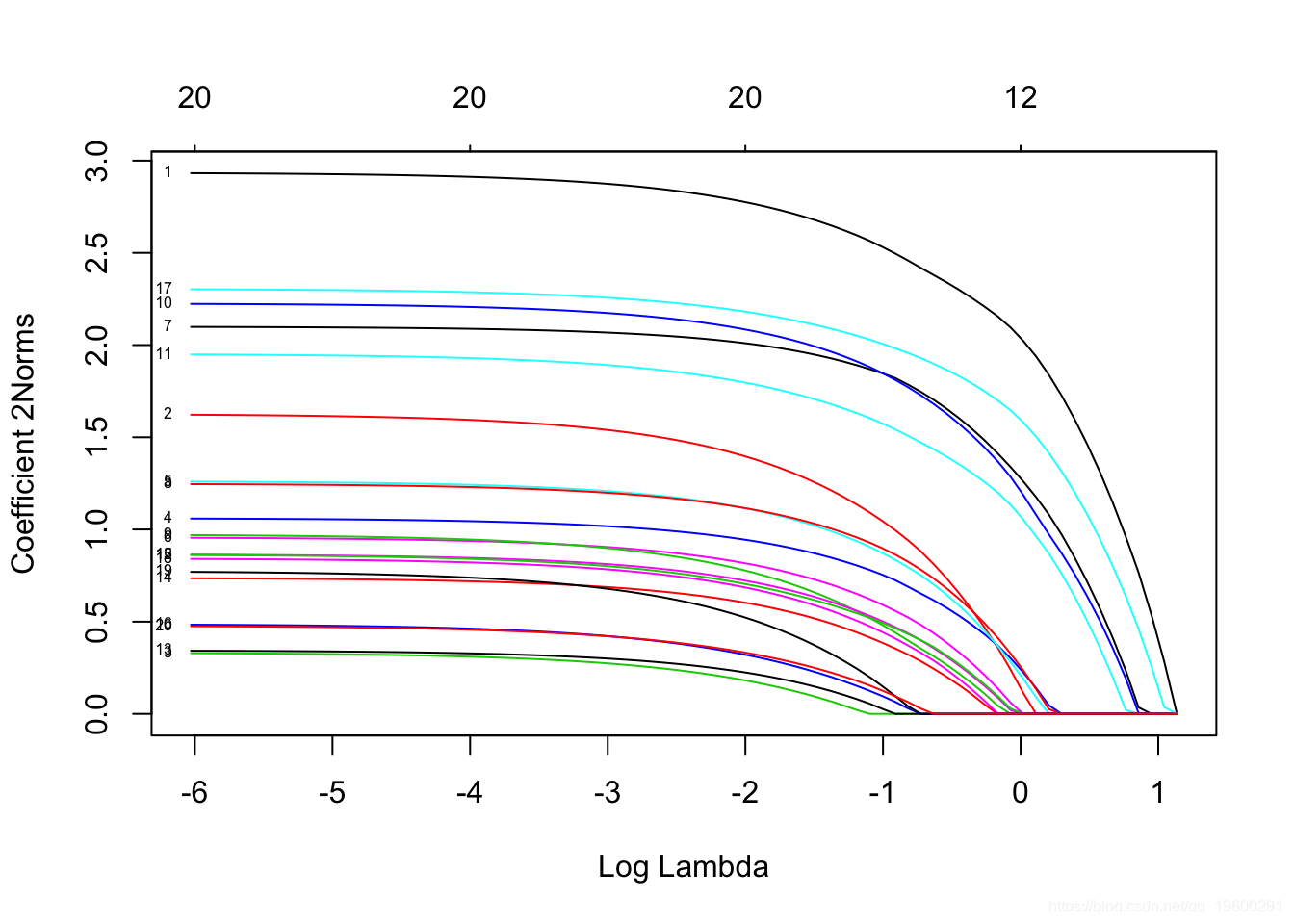
注意我们设置了 type.coef = "2norm"。在此设置下,每个变量绘制一条曲线,其值等于ℓ2范数。默认设置为 type.coef = "coef",其中为每个因变量创建一个系数图。
通过使用该函数coef ,我们可以提取要求的λ值的系数, 并通过进行预测 。
## , , 1
##
## y1 y2 y3 y4
## [1,] -4.7106 -1.1635 0.6028 3.741
## [2,] 4.1302 -3.0508 -1.2123 4.970
## [3,] 3.1595 -0.5760 0.2608 2.054
## [4,] 0.6459 2.1206 -0.2252 3.146
## [5,] -1.1792 0.1056 -7.3353 3.248
##
## , , 2
##
## y1 y2 y3 y4
## [1,] -4.6415 -1.2290 0.6118 3.780
## [2,] 4.4713 -3.2530 -1.2573 5.266
## [3,] 3.4735 -0.6929 0.4684 2.056
## [4,] 0.7353 2.2965 -0.2190 2.989
## [5,] -1.2760 0.2893 -7.8259 3.205预测结果保存在三维数组中,其中前两个维是每个因变量的预测矩阵,第三个维表示因变量。
我们还可以进行k折交叉验证。
我们绘制结果 cv.glmnet 对象“ cvmfit”。
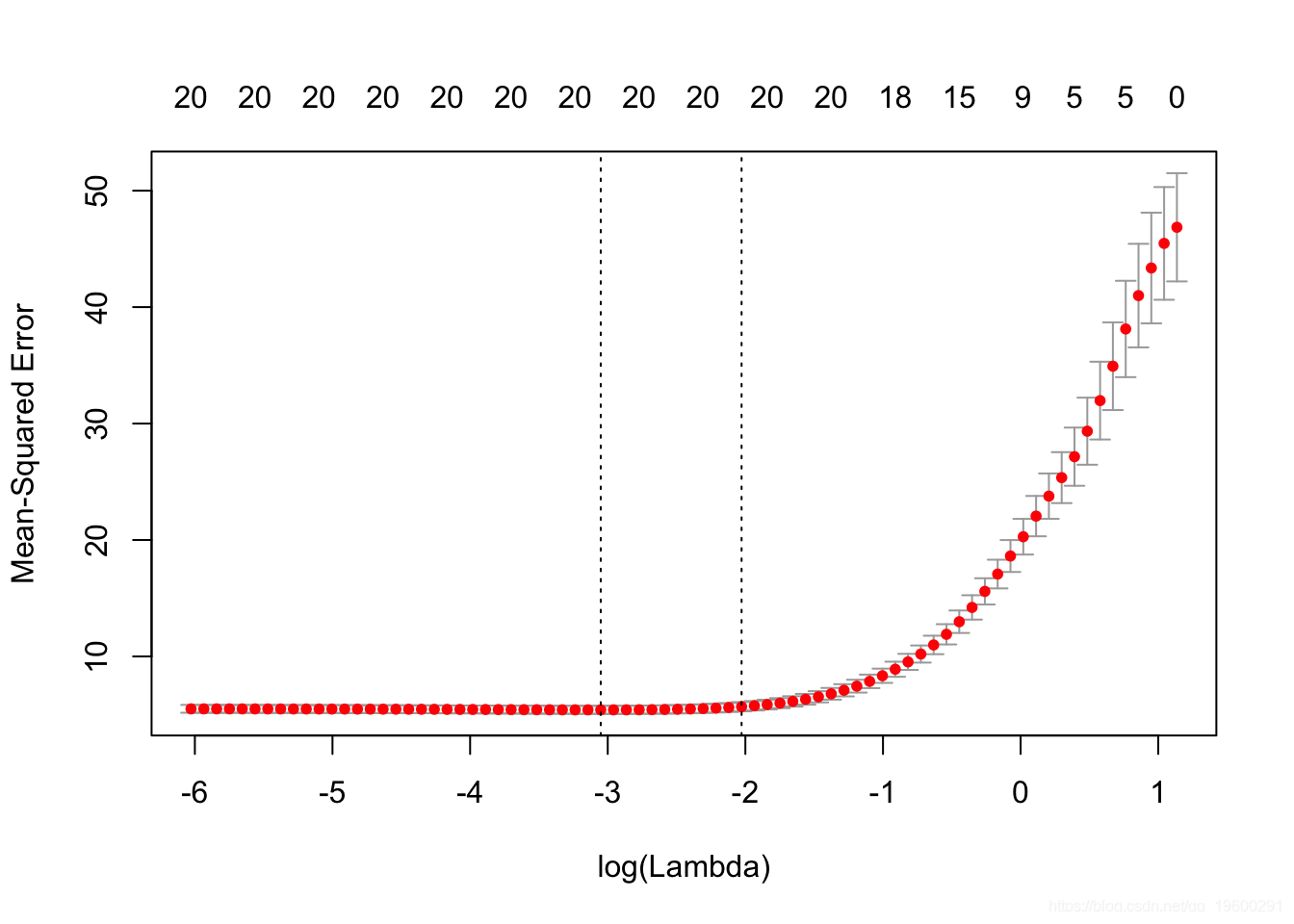
显示选定的λ最佳值
cvmfit$lambda.min## [1] 0.04732cvmfit$lambda.1se## [1] 0.1317逻辑回归
当因变量是分类的时,逻辑回归是另一个广泛使用的模型。如果有两个可能的结果,则使用二项式分布,否则使用多项式。
二项式模型
对于二项式模型,假设因变量的取值为G = {1,2} 。表示yi = I(gi = 1)。我们建模

可以用以下形式写

惩罚逻辑回归的目标函数使用负二项式对数似然

我们的算法使用对数似然的二次逼近,然后对所得的惩罚加权最小二乘问题进行下降。这些构成了内部和外部循环。
出于说明目的,我们 从数据文件加载预生成的输入矩阵 x 和因变量 y。
对于二项式逻辑回归,因变量y可以是两个级别的因子,也可以是计数或比例的两列矩阵。
glmnet 二项式回归的其他可选参数与正态分布的参数 几乎相同。不要忘记将family 选项设置 为“ binomial”。
fit = glmnet(x, y, family = "binomial")像以前一样,我们可以输出和绘制拟合的对象,提取特定λ处的系数,并进行预测。
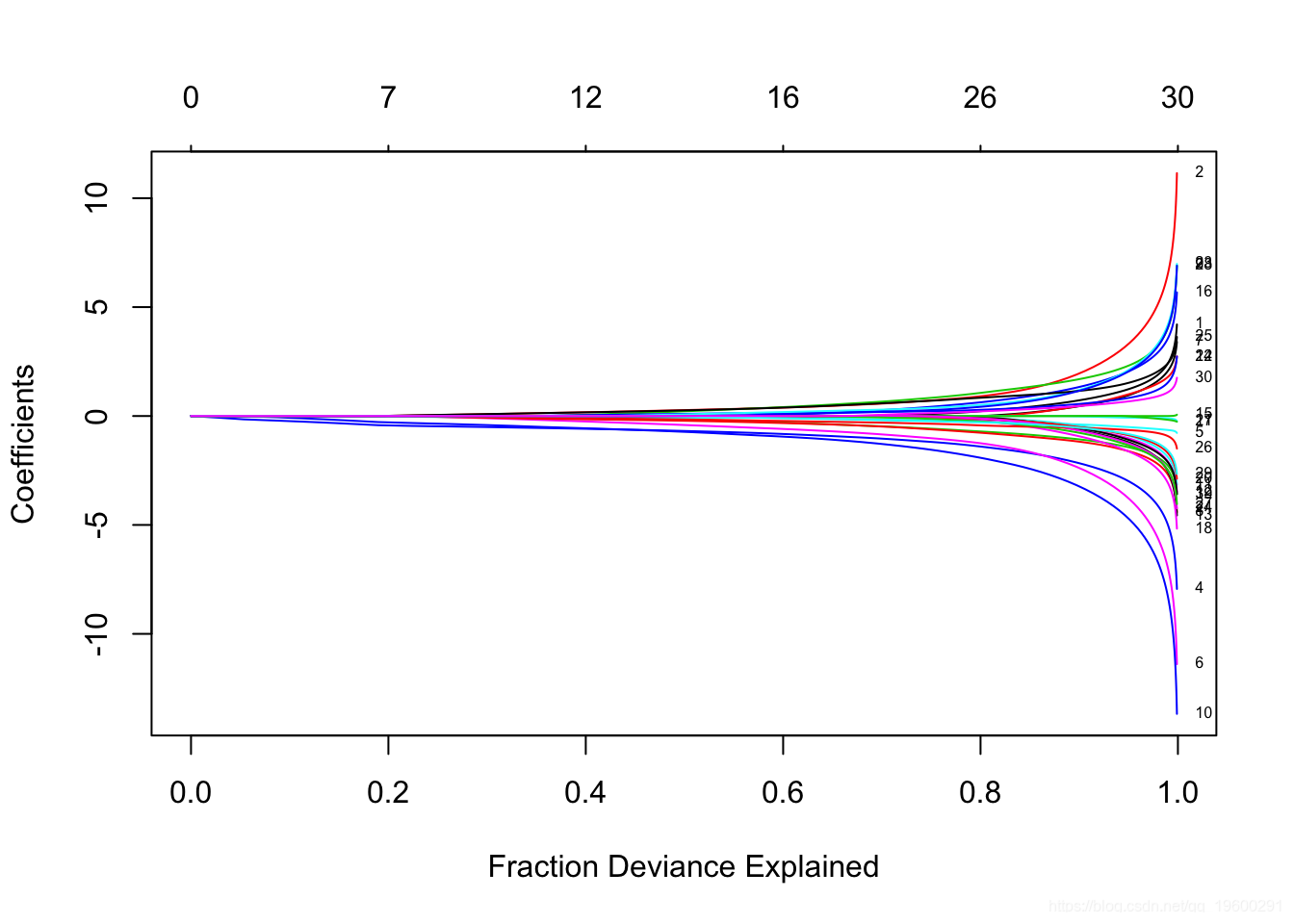
逻辑回归略有不同,主要体现在选择上 type。“链接”和“因变量”不等价,“类”仅可用于逻辑回归。总之,*“链接”给出了线性预测变量
- “因变量”给出合适的概率
- “类别”产生对应于最大概率的类别标签。
- “系数”计算值为的系数
s
在下面的示例中,我们在λ=0.05,0.01的情况下对类别标签进行了预测。
## 1 2
## [1,] "0" "0"
## [2,] "1" "1"
## [3,] "1" "1"
## [4,] "0" "0"
## [5,] "1" "1"对于逻辑回归,type.measure:
- “偏差”使用实际偏差。
- “ mae”使用平均绝对误差。
- “class”给出错误分类错误。
- “ auc”(仅适用于两类逻辑回归)给出了ROC曲线下的面积。
例如,
它使用分类误差作为10倍交叉验证的标准。
我们绘制对象并显示λ的最佳值。
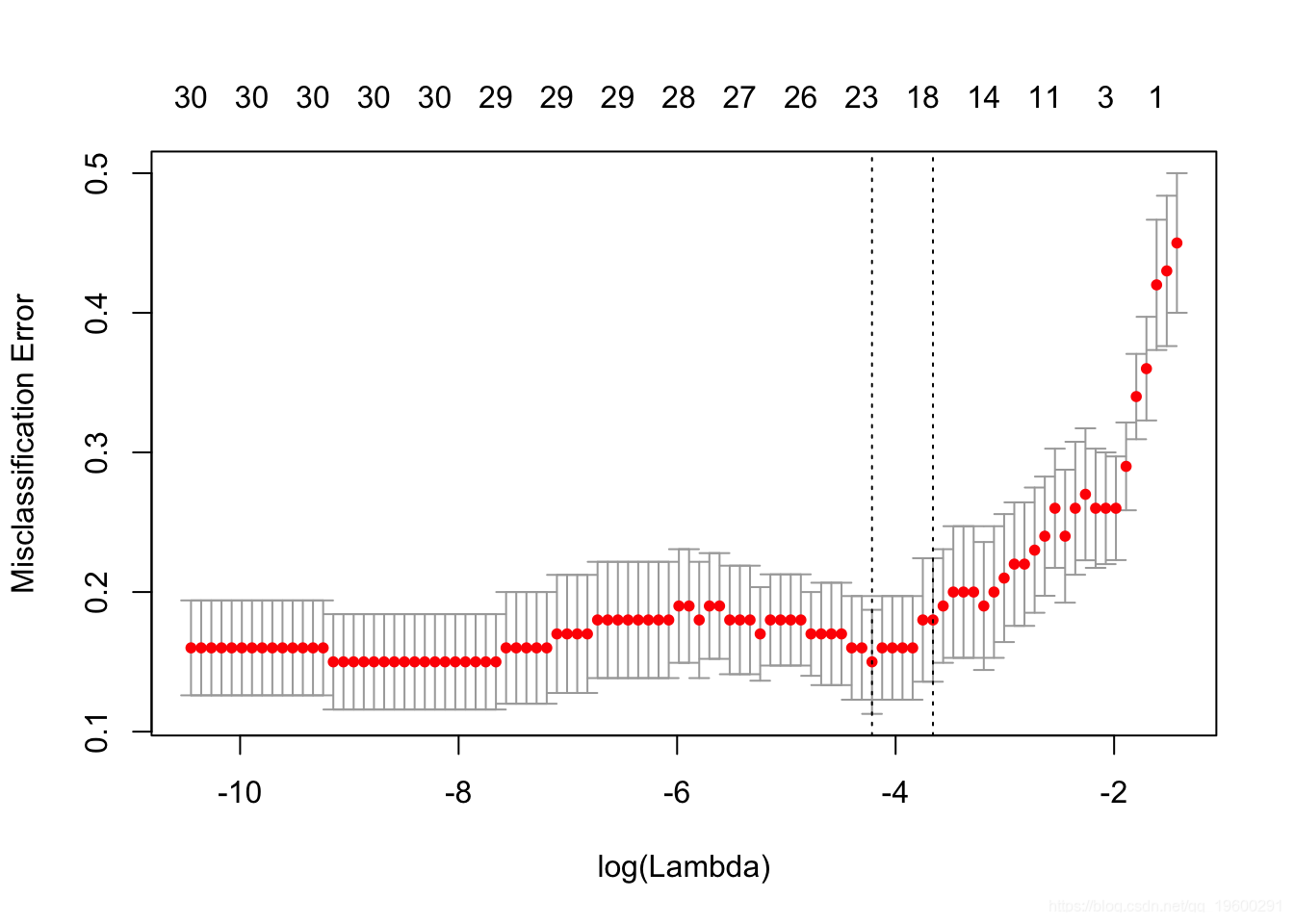
cvfit$lambda.min## [1] 0.01476cvfit$lambda.1se## [1] 0.02579coef 并且 predict 类似于正态分布案例,因此我们省略了细节。我们通过一些例子进行回顾。
## 31 x 1 sparse Matrix of class "dgCMatrix"
## 1
## (Intercept) 0.24371
## V1 0.06897
## V2 0.66252
## V3 -0.54275
## V4 -1.13693
## V5 -0.19143
## V6 -0.95852
## V7 .
## V8 -0.56529
## V9 0.77454
## V10 -1.45079
## V11 -0.04363
## V12 -0.06894
## V13 .
## V14 .
## V15 .
## V16 0.36685
## V17 .
## V18 -0.04014
## V19 .
## V20 .
## V21 .
## V22 0.20882
## V23 0.34014
## V24 .
## V25 0.66310
## V26 -0.33696
## V27 -0.10570
## V28 0.24318
## V29 -0.22445
## V30 0.11091如前所述,此处返回的结果仅针对因子因变量的第二类。
## 1
## [1,] "0"
## [2,] "1"
## [3,] "1"
## [4,] "0"
## [5,] "1"
## [6,] "0"
## [7,] "0"
## [8,] "0"
## [9,] "1"
## [10,] "1"多项式模型
对于多项式模型,假设因变量变量的K级别为G = {1,2,…,K}。在这里我们建模

设Y为N×K指标因变量矩阵,元素yiℓ= I(gi =ℓ)。然后弹性网惩罚的负对数似然函数变为

β是系数的p×K矩阵。βk指第k列(对于结果类别k),βj指第j行(变量j的K个系数的向量)。最后一个惩罚项是||βj|| q ,我们对q有两个选择:q∈{1,2}。当q = 1时,这是每个参数的套索惩罚。当q = 2时,这是对特定变量的所有K个系数的分组套索惩罚,这使它们在一起全为零或非零。
对于多项式情况,用法类似于逻辑回归,我们加载一组生成的数据。
glmnet 除少数情况外,多项式逻辑回归中的可选参数 与二项式回归基本相似。
多项式回归的一个特殊选项是 type.multinomial,如果允许,则允许使用分组的套索罚分 type.multinomial = "grouped"。这将确保变量的多项式系数全部一起输入或输出,就像多元因变量一样。
我们绘制结果。
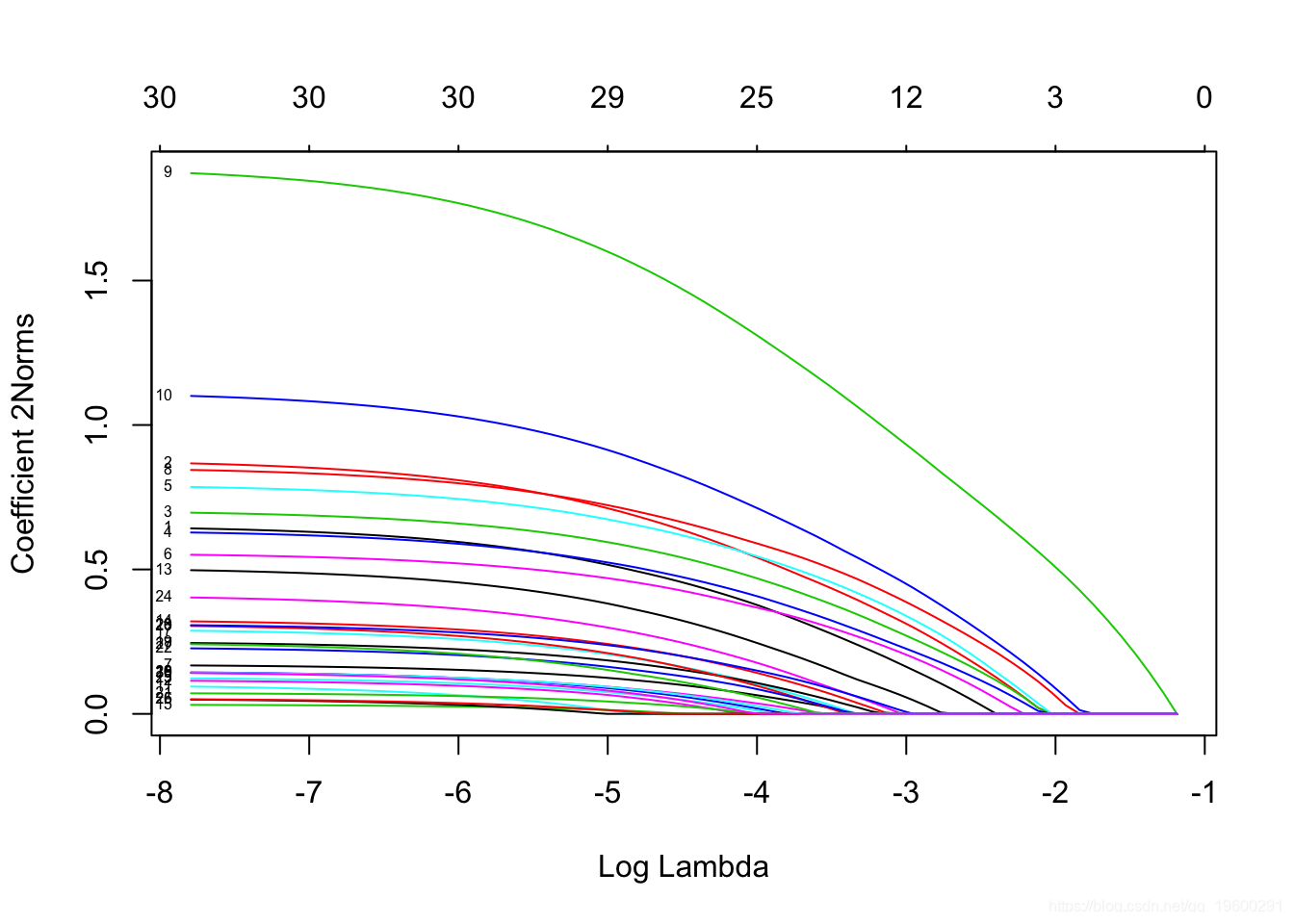
我们还可以进行交叉验证并绘制返回的对象。
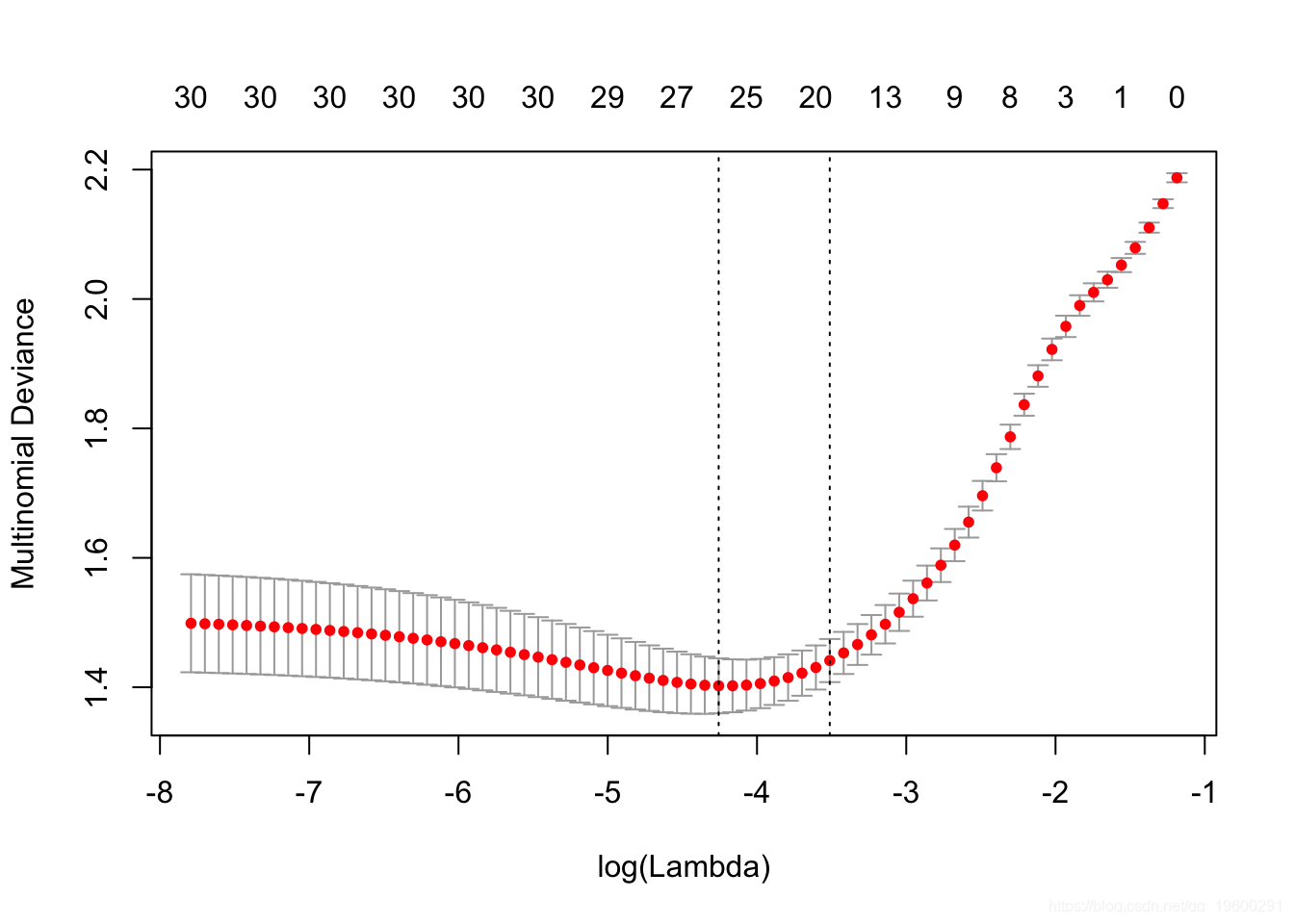
预测最佳选择的λ:
## 1
## [1,] "3"
## [2,] "2"
## [3,] "2"
## [4,] "1"
## [5,] "1"
## [6,] "3"
## [7,] "3"
## [8,] "1"
## [9,] "1"
## [10,] "2"泊松模型
Poisson回归用于在假设Poisson误差的情况下对计数数据进行建模,或者在均值和方差成比例的情况下使用非负数据进行建模。泊松也是指数分布族的成员。我们通常以对数建模:![]() 。
。
给定观测值![]() 的对数似然
的对数似然

和以前一样,我们优化了惩罚对数:

Glmnet使用外部牛顿循环和内部加权最小二乘循环(如逻辑回归)来优化此标准。
首先,我们加载一组泊松数据。
再次,绘制系数。
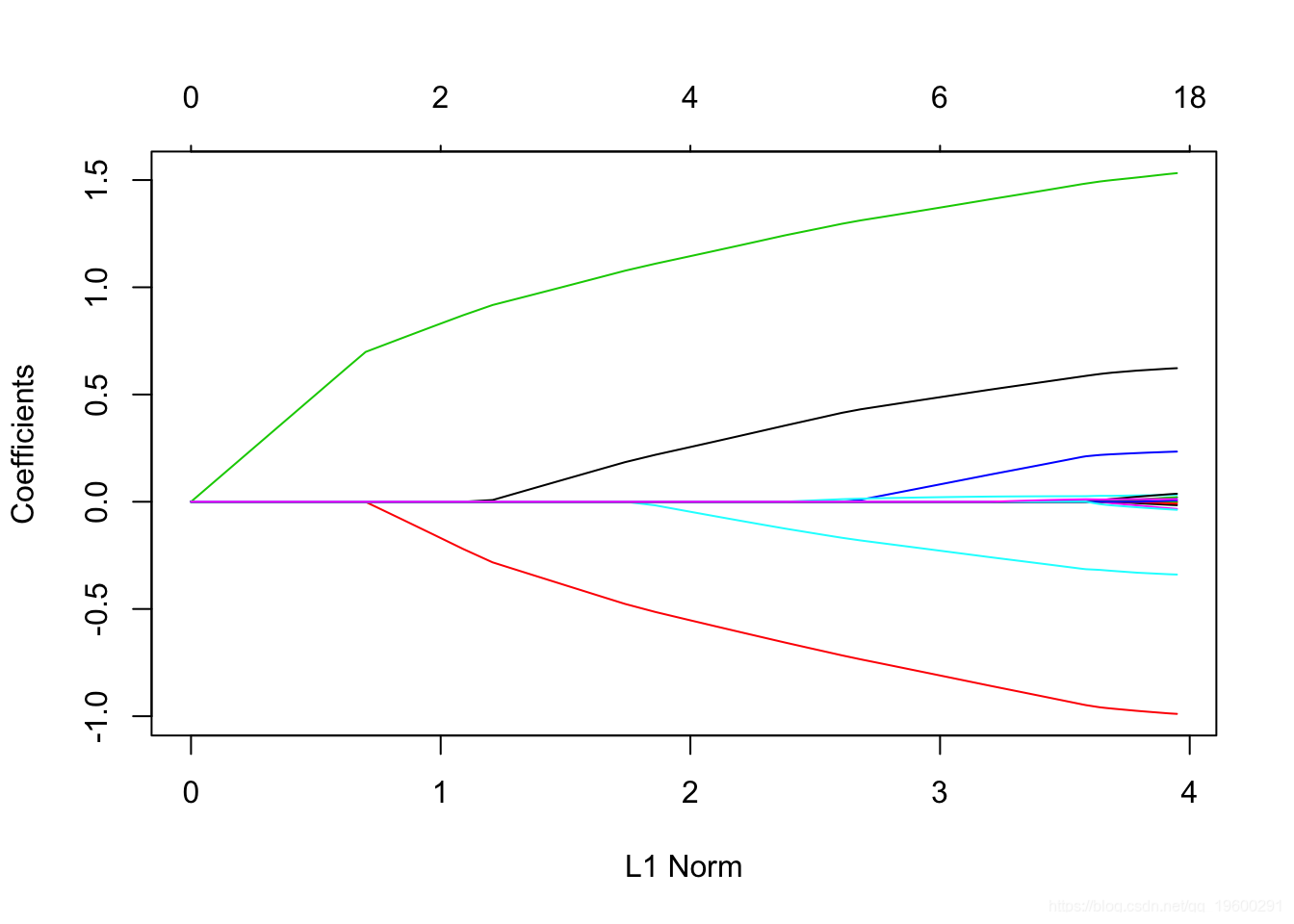
像以前一样,我们可以 分别使用coef 和 提取系数并在特定的λ处进行预测 predict。
例如,我们可以
## 21 x 1 sparse Matrix of class "dgCMatrix"
## 1
## (Intercept) 0.61123
## V1 0.45820
## V2 -0.77061
## V3 1.34015
## V4 0.04350
## V5 -0.20326
## V6 .
## V7 .
## V8 .
## V9 .
## V10 .
## V11 .
## V12 0.01816
## V13 .
## V14 .
## V15 .
## V16 .
## V17 .
## V18 .
## V19 .
## V20 .## 1 2
## [1,] 2.4944 4.4263
## [2,] 10.3513 11.0586
## [3,] 0.1180 0.1782
## [4,] 0.9713 1.6829
## [5,] 1.1133 1.9935我们还可以使用交叉验证来找到最佳的λ,从而进行推断。
选项几乎与正态族相同,不同之处在于 type.measure*,“ mse”代表均方误差*,“ mae”代表均值绝对误差。
我们可以绘制 cv.glmnet 对象。
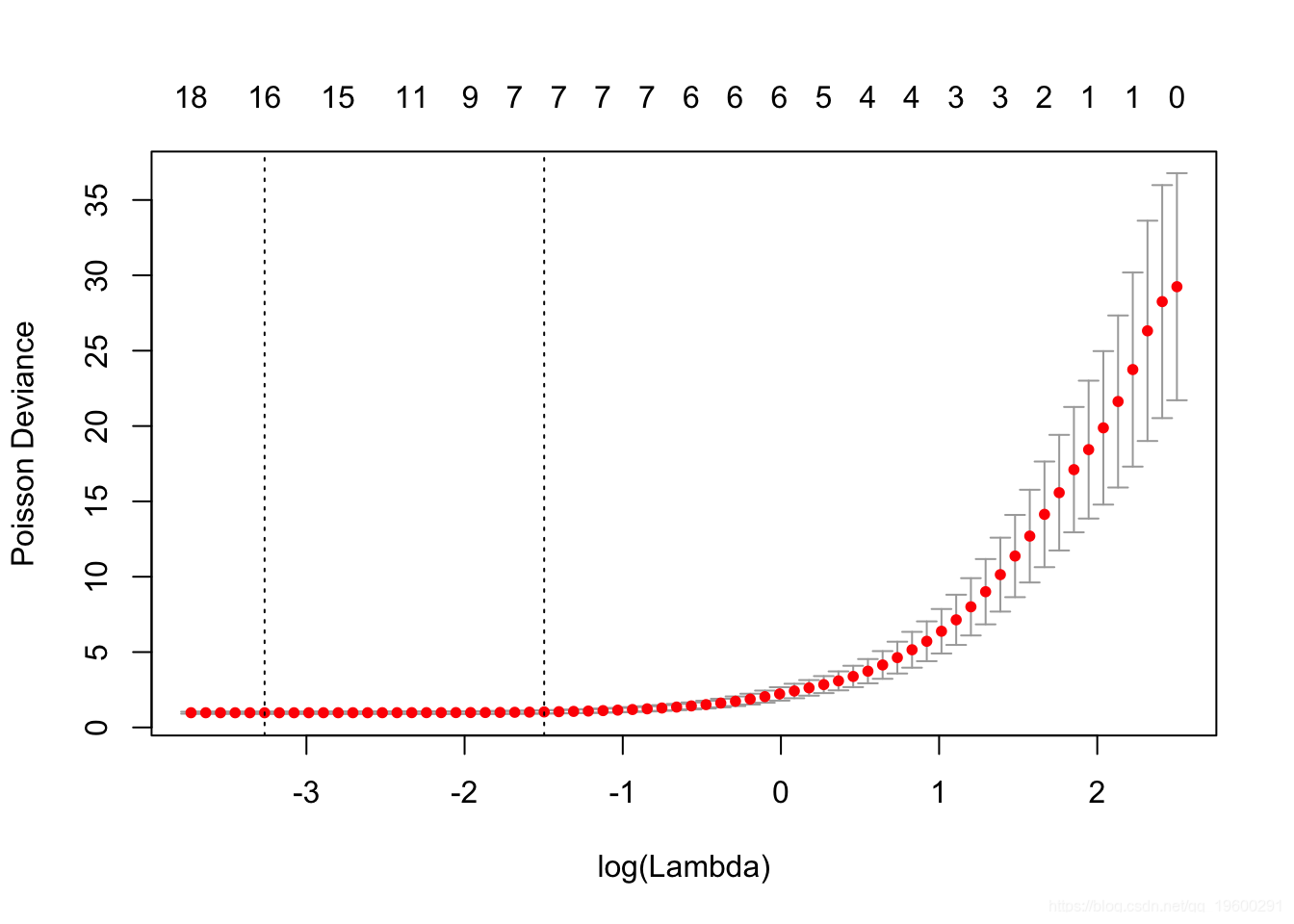
我们还可以显示最佳的λ和相应的系数。
## 21 x 2 sparse Matrix of class "dgCMatrix"
## 1 2
## (Intercept) 0.031263 0.18570
## V1 0.619053 0.57537
## V2 -0.984550 -0.93212
## V3 1.525234 1.47057
## V4 0.231591 0.19692
## V5 -0.336659 -0.30469
## V6 0.001026 .
## V7 -0.012830 .
## V8 . .
## V9 . .
## V10 0.015983 .
## V11 . .
## V12 0.030867 0.02585
## V13 -0.027971 .
## V14 0.032750 .
## V15 -0.005933 .
## V16 0.017506 .
## V17 . .
## V18 0.004026 .
## V19 -0.033579 .
## V20 0.012049 0.00993Cox模型
Cox比例风险模型通常用于研究预测变量与生存时间之间的关系。
Cox比例风险回归模型,它不是直接考察![]() 与X的关系,而是用
与X的关系,而是用![]() 作为因变量,模型的基本形式为:
作为因变量,模型的基本形式为:

式中,![]() 为自变量的偏回归系数,它是须从样本数据作出估计的参数;
为自变量的偏回归系数,它是须从样本数据作出估计的参数;![]() 是当X向量为0时,
是当X向量为0时,![]() 的基准危险率,它是有待于从样本数据作出估计的量。简称为Cox回归模型。
的基准危险率,它是有待于从样本数据作出估计的量。简称为Cox回归模型。
由于Cox回归模型对![]() 未作任何假定,因此Cox回归模型在处理问题时具有较大的灵活性;另一方面,在许多情况下,我们只需估计出参数
未作任何假定,因此Cox回归模型在处理问题时具有较大的灵活性;另一方面,在许多情况下,我们只需估计出参数![]() (如因素分析等),即使在
(如因素分析等),即使在![]() 未知的情况下,仍可估计出参数
未知的情况下,仍可估计出参数![]() 。这就是说,Cox回归模型由于含有
。这就是说,Cox回归模型由于含有![]() ,因此它不是完全的参数模型,但仍可根据公式(1)作出参数
,因此它不是完全的参数模型,但仍可根据公式(1)作出参数![]() 的估计,故Cox回归模型属于半参数模型。
的估计,故Cox回归模型属于半参数模型。
公式可以转化为:![]()
我们使用一组预先生成的样本数据。用户可以加载自己的数据并遵循类似的过程。在这种情况下,x必须是协变量值的n×p矩阵-每行对应一个患者,每列对应一个协变量。y是一个n×2矩阵。
## time status
## [1,] 1.76878 1
## [2,] 0.54528 1
## [3,] 0.04486 0
## [4,] 0.85032 0
## [5,] 0.61488 1Surv 包中的 函数 survival 可以创建这样的矩阵。
我们计算默认设置下的求解路径。
绘制系数。

提取特定值λ处的系数。
## 30 x 1 sparse Matrix of class "dgCMatrix"
## 1
## V1 0.37694
## V2 -0.09548
## V3 -0.13596
## V4 0.09814
## V5 -0.11438
## V6 -0.38899
## V7 0.24291
## V8 0.03648
## V9 0.34740
## V10 0.03865
## V11 .
## V12 .
## V13 .
## V14 .
## V15 .
## V16 .
## V17 .
## V18 .
## V19 .
## V20 .
## V21 .
## V22 .
## V23 .
## V24 .
## V25 .
## V26 .
## V27 .
## V28 .
## V29 .
## V30 .函数 cv.glmnet 可用于计算Cox模型的k折交叉验证。
拟合后,我们可以查看最佳λ值和交叉验证的误差图,帮助评估我们的模型。
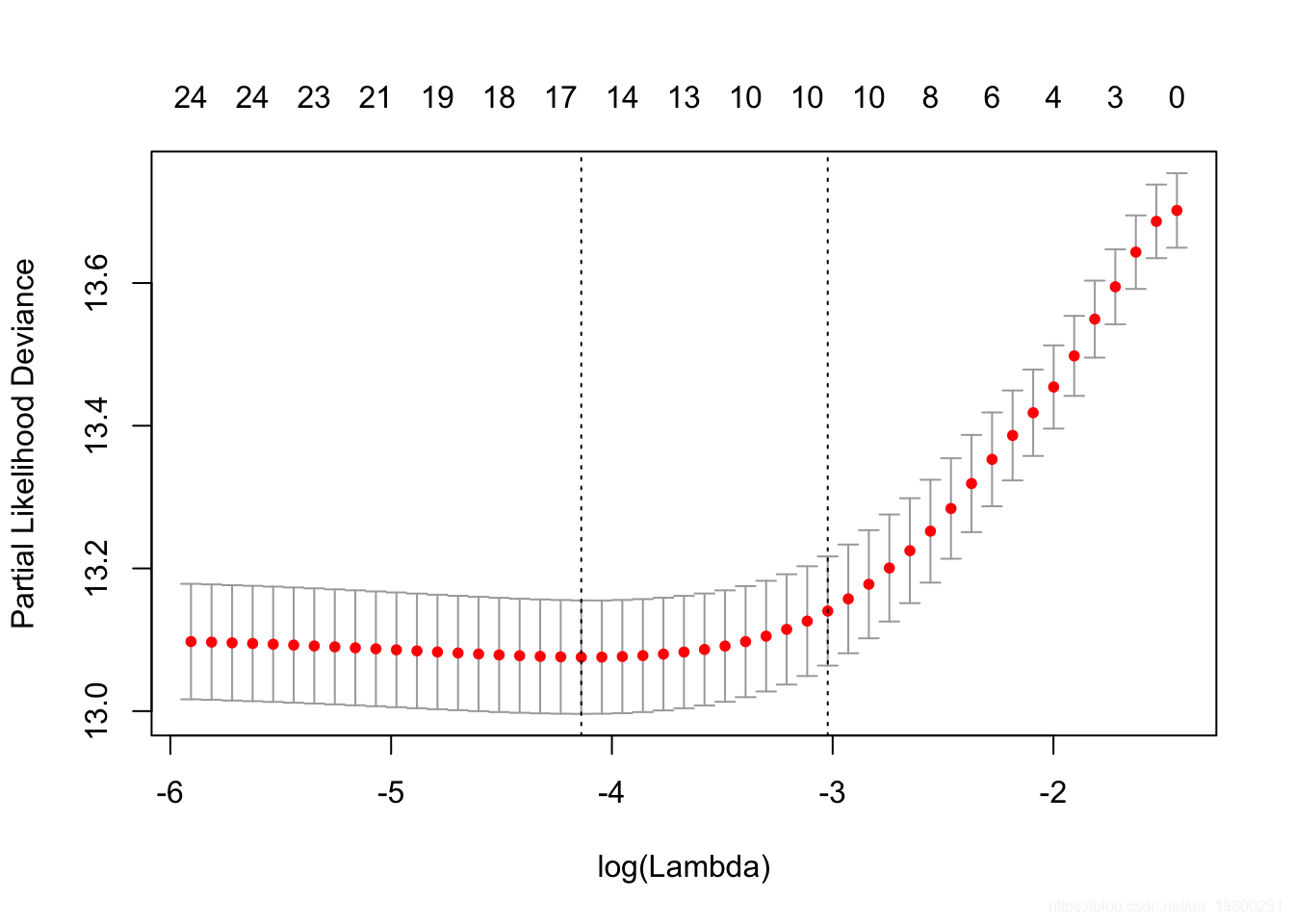
如前所述,图中的左垂直线向我们显示了CV误差曲线达到最小值的位置。右边的垂直线向我们展示了正则化的模型,其CV误差在最小值的1个标准偏差之内。我们还提取了最优λ。
cvfit$lambda.min## [1] 0.01594cvfit$lambda.1se## [1] 0.04869我们可以检查模型中的协变量并查看其系数。
index.min## [1] 0.491297 -0.174601 -0.218649 0.175112 -0.186673 -0.490250 0.335197
## [8] 0.091587 0.450169 0.115922 0.017595 -0.018365 -0.002806 -0.001423
## [15] -0.023429 0.001688 -0.008236coef.min## 30 x 1 sparse Matrix of class "dgCMatrix"
## 1
## V1 0.491297
## V2 -0.174601
## V3 -0.218649
## V4 0.175112
## V5 -0.186673
## V6 -0.490250
## V7 0.335197
## V8 0.091587
## V9 0.450169
## V10 0.115922
## V11 .
## V12 .
## V13 0.017595
## V14 .
## V15 .
## V16 .
## V17 -0.018365
## V18 .
## V19 .
## V20 .
## V21 -0.002806
## V22 -0.001423
## V23 .
## V24 .
## V25 -0.023429
## V26 .
## V27 0.001688
## V28 .
## V29 .
## V30 -0.008236稀疏矩阵
我们的程序包支持稀疏的输入矩阵,该矩阵可以高效地存储和操作大型矩阵,但只有少数几个非零条目。
我们加载一组预先创建的样本数据。
加载100 * 20的稀疏矩阵和 y因向量。
## [1] "dgCMatrix"
## attr(,"package")
## [1] "Matrix"我们可以像以前一样拟合模型。
fit = glmnet(x, y)进行交叉验证并绘制结果对象。

预测新输入矩阵 。例如,
## 1
## [1,] 0.3826
## [2,] -0.2172
## [3,] -1.6622
## [4,] -0.4175
## [5,] -1.3941参考文献
Jerome Friedman, Trevor Hastie and Rob Tibshirani. (2008).
Regularization Paths for Generalized Linear Models via Coordinate Descent

 Python房价数据预测:StackingCVRegressor集成学习、Lasso、ElasticNet、XGBoost、LightGBM模型与特征工程可视化
Python房价数据预测:StackingCVRegressor集成学习、Lasso、ElasticNet、XGBoost、LightGBM模型与特征工程可视化 Python电影票房预测模型研究——贝叶斯岭回归Ridge、决策树、Adaboost、KNN分析猫眼豆瓣数据
Python电影票房预测模型研究——贝叶斯岭回归Ridge、决策树、Adaboost、KNN分析猫眼豆瓣数据 Python员工数据人力流失预测:ADASYN采样CatBoost算法、LASSO特征选择与动态不平衡处理及多模型对比研究
Python员工数据人力流失预测:ADASYN采样CatBoost算法、LASSO特征选择与动态不平衡处理及多模型对比研究



