PLS-DA (Partial Least Squares Discriminant Analysis) 是一种多变量统计分析方法,常用于处理具有多个预测变量和多个响应变量的数据。
在本文中,我们帮助客户使用了PLS-DA方法来挖掘两个疾病的不同中医分组方式下存在差异的指标。
首先,我们有两个Excel文件,分别是患者的证素数据。
可下载资源
每一列代表一位患者的多个数据,不同颜色代表了不同的分组。我们想要通过PLS-DA挖掘不同组别患者间存在差异的指标。
两个EXCEL分别是患者的证素的数据,由于是评分性质的,所以都是不连续的数字。
每一列代表一位患者的多个数据,不同颜色代表了不同的分组,想通过PLS-DA挖掘下不同组别患者间存在差异的指标有哪些。2个EXCEL是分开的2个疾病,每个疾病下包含不同中医的分组方式,主要想挖掘下不同中医分组方式下存在差异的指标。一方面需要找到这些存在差异的指标,每一列代表一位患者的多个数据,不同颜色代表了不同的分组。
数据1
在R语言中,我们首先将数据导入并进行预处理。我们使用read.csv函数将数据1导入,并将不需要的列删除。然后,我们使用na.omit函数删除含有缺失值的行。最后,我们为每个患者指定一个组别,分别为A、B、C、D、E和F。
data=read.csv("数据1.csv") X=data X=X[,-53] #分别设置组别和指标 X=na.omit(X) Y=c(rep("A",29),rep("B",19),rep("C",27),rep("D", 8),rep("E",9),rep("F",4) )
进行PLS-DA模型的建立
接下来,我们使用PLS-DA建立模型。建立PLS-DA模型,并将数据集和组别变量作为输入。建立模型后,我们可以查看不同组别分别有哪些指标,以及哪些指标之间存在显著的差异。
tIndiv(plsda.breast,
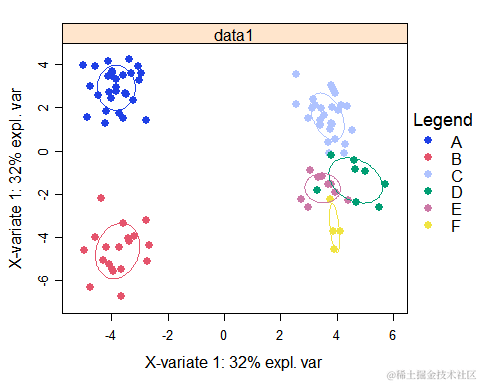
从结果中可以看到不同组别分别有哪些指标,以及哪些指标之间存在显著的差异?
从图中可以看到,分组a和分组b之间存在显著的差异,分组cdef之间的差异较小,分组a分组b和分组cdef间均存在显著差异
指示变量矩阵
st(t(plsda.breast$ind.mat))

从指示变量矩阵的结果来看,a的特征向量和b的特征向量之间存在显著差异,而cdef之间的差异较小
随时关注您喜欢的主题
数据2
接下来,我们导入数据2,并进行相似的分析步骤。首先,我们使用read.csv函数将数据2导入。然后,我们建立PLS-DA模型,并使用div函数查看不同组别分别有哪些指标,以及哪些指标之间存在显著的差异。
进行PLS-DA模型的建立
div(plsda.breast,
ellipse = TRUE
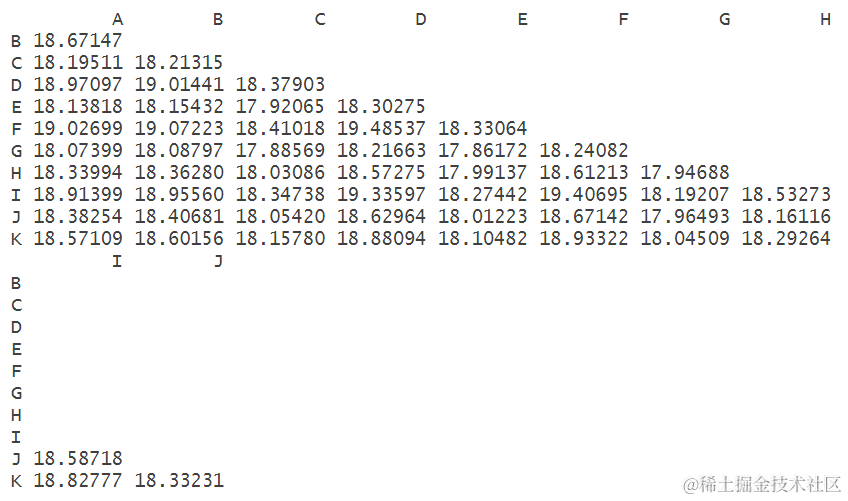
指示变量矩阵
ist(t(plsda.breast$i
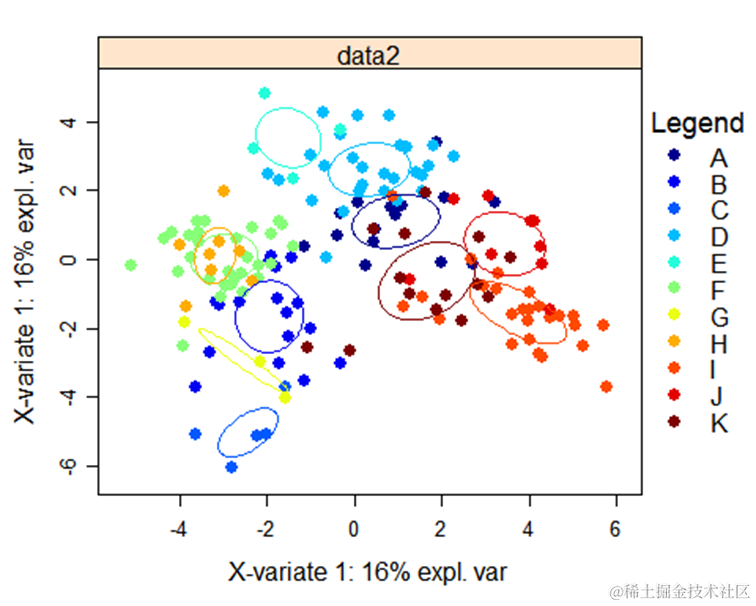
从结果中可以看到不同组别分别有哪些指标,以及哪些指标之间存在显著的差异?
从图中可以看到,分组GHEC之间的差异较小,分组ABDFIJK之间差异较小,这两类间均存在显著差异。
从指示变量矩阵的结果来看, GHEC特征向量之间的差异较小距离也较小,分组ABDFIJK之间差异较小距离也较小,这两类间均存在显著差异。
可下载资源
关于作者
Kaizong Ye是拓端研究室(TRL)的研究员。在此对他对本文所作的贡献表示诚挚感谢,他在上海财经大学完成了统计学专业的硕士学位,专注人工智能领域。擅长Python.Matlab仿真、视觉处理、神经网络、数据分析。
本文借鉴了作者最近为《R语言数据分析挖掘必知必会 》课堂做的准备。
非常感谢您阅读本文,如需帮助请联系我们!

 【专题】人工智能行业C_Suite全球AI指标报告报告合集PDF分享(附原数据表)
【专题】人工智能行业C_Suite全球AI指标报告报告合集PDF分享(附原数据表) R语言主成分pca、因子分析、聚类对地区经济研究分析重庆市经济指标
R语言主成分pca、因子分析、聚类对地区经济研究分析重庆市经济指标 R语言 PCA(主成分分析),CA(对应分析)夫妻职业差异和马赛克图可视化
R语言 PCA(主成分分析),CA(对应分析)夫妻职业差异和马赛克图可视化


