我们使用R中的igraph包,产生了网络的图形。
但是很难将这些图表放到演讲和文章中,因为图表很难根据需要定制。
使用igraph中的绘图功能可以得到你想要的结果,但用ggplot对工作更有帮助。所以本文探索了一种在ggplot中创建igraph绘图的方法。
网络是由一些紧密相连的节点组成的,并且根据不同节点之间连接的紧密程度,网络也可视为由不同簇组成。簇内的节点之间有着更为紧密的连接,不同簇之间的连接则相对稀疏。这种簇被称为网络中的社区结构(community structure)。
由此衍生出来的社区发现(community detection)算法用来发现网络中的社区结构,这类算法包括 Louvain 算法、Girvan-Newman 算法以及 Bron-Kerbosch 算法等。
首先,该库可以实现以下几种社区发现算法:
-
Louvain 算法
-
Girvan-Newman 算法
-
层次聚类
-
谱聚类
-
Bron-Kerbosch 算法
算法详解
Louvain 算法
louvain_method(adj_matrix : numpy.ndarray, n : int = None) -> list
该算法来源于文章《Fast unfolding of communities in large networks》,简称为 Louvian。
作为一种基于模块度(Modularity)的社区发现算法,Louvain 算法在效率和效果上都表现比较好,并且能够发现层次性的社区结构,其优化的目标是最大化整个图属性结构(社区网络)的模块度。
Louvain 算法对最大化图模块性的社区进行贪婪搜索。如果一个图具有高密度的群体内边缘和低密度的群体间边缘,则称之为模图。
Girvan-Newman 算法
girvan_newman(adj_matrix : numpy.ndarray, n : int = None) -> list
该算法来源于文章《Community structure in social and biological networks》。
Girvan-Newman 算法迭代删除边以创建更多连接的组件。每个组件都被视为一个 community,当模块度不能再增加时,算法停止去除边缘。
层次聚类
hierarchical_clustering(adj_matrix : numpy.ndarray, metric : str = "cosine", linkage : str = "single", n : int = None) -> list
层次聚类实现了一种自底向上、分层的聚类算法。每个节点从自己 的社区开始,然后,随着层次结构的建立,最相似的社区被合并。社区会一直被合并,直到在模块度方面没有进一步的进展。
谱聚类
spectral_clustering(adj_matrix : numpy.ndarray, k : int) -> list
这种类型的算法假定邻接矩阵的特征值包含有关社区结构的信息。
Bron-Kerbosch 算法
bron_kerbosch(adj_matrix : numpy.ndarray, pivot : bool = False) -> list
Bron-Kerbosch 算法实现用于最大团检测(maximal clique detection)。图中的最大团是形成一个完整图的节点子集,如果向该子集中添加其他节点,则它将不再完整。将最大团视为社区是合理的,因为团是图中连接最紧密的节点群。因为一个节点可以是多个社区的成员,所以该算法有时会识别重叠的社区。
igraph图
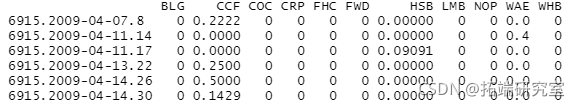
首先,我将带入数据,这是一个物种相对丰度的矩阵。列是物种,每行是一个观测值。下面是数据的浏览
head(data.wide)
加载igraph库并运行生成网络的前几个步骤
library(igraph) all <- bipartite.projection(inc)
绘制这两幅图产生的图形还可以,但并不美观。
op <- par(mfrow = c(1, 2)) plot(obs, layout = layout.fruchterman.reingold, edge.color = "black") par(op)
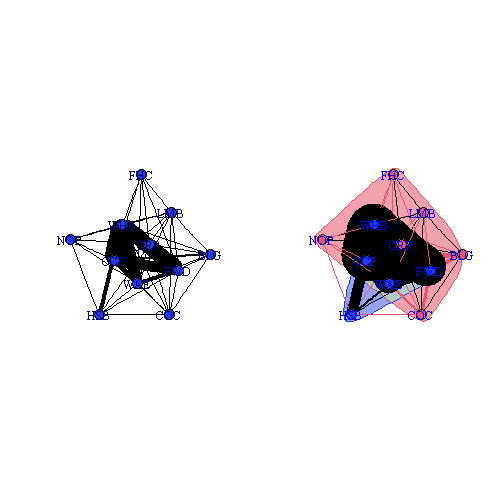
在ggplot中创建图形
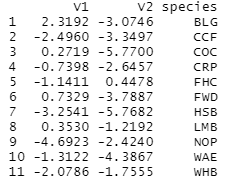
首先让我们提取数据,产生左边的网络基本图。GGPLOT需要数据为数据框,所以提取数据并将其转换为数据框
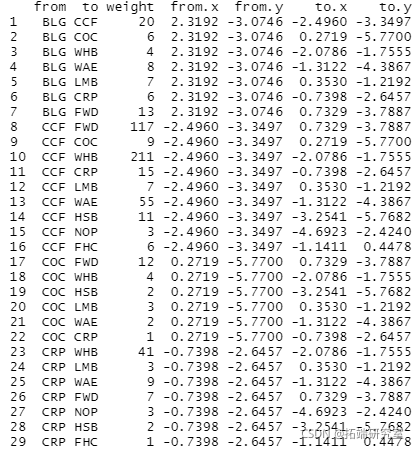
species <- colnames(wide2) ## 添加物种代码 df ## 显示每个节点的x(V1)和y(V2)坐标。
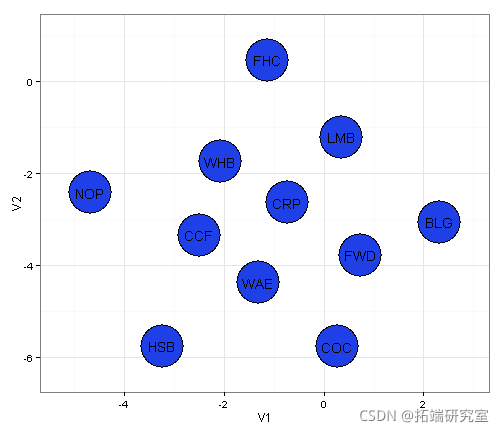
现在我们有了图中所有节点的坐标,我们可以在ggplot中绘制了
library(ggplot2) ggplot() + geom_point( color="black") + # 在节点周围添加一个黑色的边框 geom_text( label=species ) + # 添加节点的标签
现在我们有了正确的节点,画出节点之间的连接。
get(obs) # 使用函数获得边信息
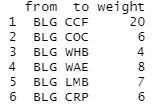
df\[match(from, species)\] # 匹配之前连接的节点数据框架中的 from 位置。 gto <- all\[match(to, specie)\] # 匹配之前连接的节点数据框中的to位置
随时关注您喜欢的主题
然后绘制
ggplot() + geom_point(color="black") + # 在节点周围添加一个黑色的边 geom_text(label=species)) + # 添加节点的标签
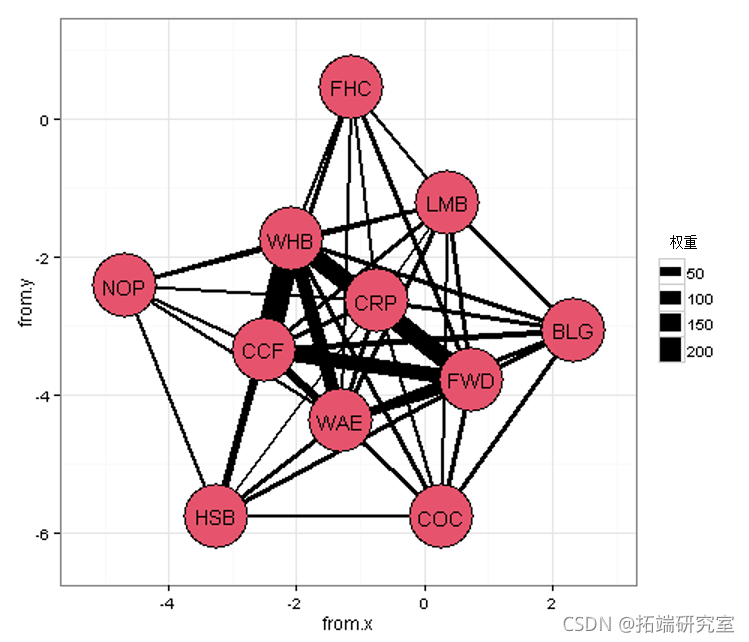
让我们弄乱主题,删除网格线和轴标签等。
ggplot() + geom_point(color="black") + # 在节点周围添加一个黑色的边 geom_text(label=species)) + # 添加节点的标签 axis.text.x = element_blank(), # 移除x轴文字 axis.text.y = element_blank(), #删除y轴文字 axis.ticks = element_blank(), # 删除轴的刻度线 axis.title.x = element_blank(), # 删除X轴标签 axis.title.y = element_blank(), # 删除y轴标签 panel.grid.major = element_blank(), #移除主要网格的标签 panel.grid.minor = element_blank(), #删除minor-grid标签
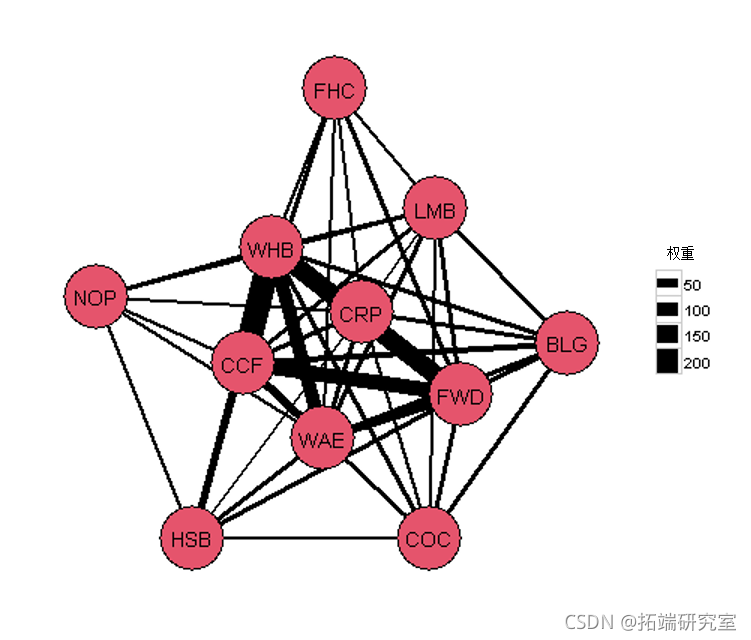
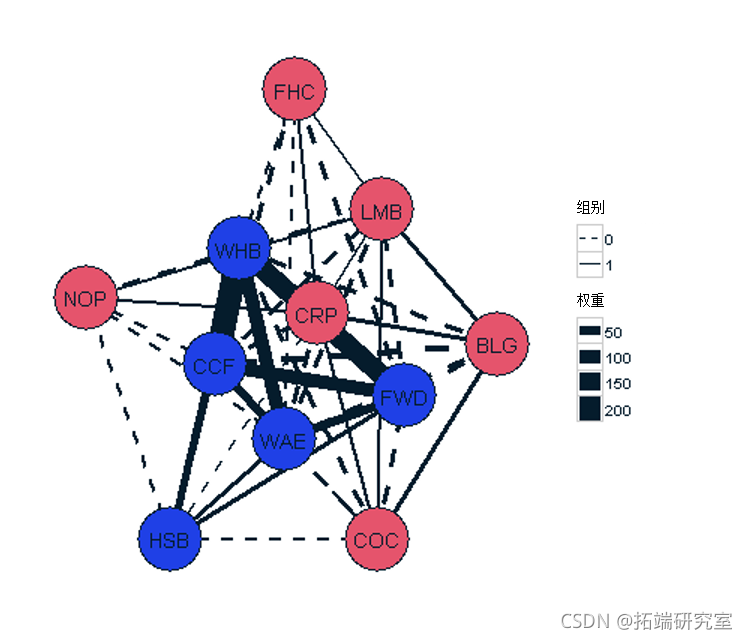
如果我们想把社区检测算法中的一些元素纳入右边的图中。我们可以把一个组中的元素变成红色,另一个组中的元素变成蓝色。
组内的连接将是一条实线,组间的连接将是一条虚线。
data.frame(sp = names, g=membership) #创建一个物种和组成员的数据框架 g\[match( from, sp )\] # 在g数据框中为from和to节点匹配组成员资格
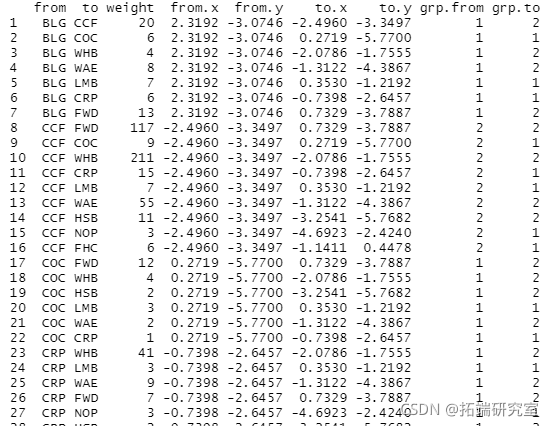
将组类型添加到节点数据框中。

grp <- group\[match( species, species)\] #
ggplot() + geom_segment(type=as.factor(type)),color="black") + # 添加线 geom_point(color="black") + # 在节点周围添加一个黑色的边界。 geom_text(label=species)) + # 添加节点的标签 theme_bw()+ # 使用ggplot的黑白主题 theme( axis.text.x = element_blank(), # 移除x轴文字 axis.text.y = element_blank(), #删除y轴文字 axis.ticks = element_blank(), # 删除轴的刻度线 axis.title.x = element_blank(), # 删除X轴标签 axis.title.y = element_blank(), # 删除y轴标签 panel.grid.major = element_blank(), #移除主要网格的标签 panel.grid.minor = element_blank(), #删除minor-grid标签
可下载资源
关于作者
Kaizong Ye是拓端研究室(TRL)的研究员。Kaizong Ye是拓端研究室(TRL)的研究员。在此对他对本文所作的贡献表示诚挚感谢,他在上海财经大学完成了统计学专业的硕士学位,专注人工智能领域。擅长Python.Matlab仿真、视觉处理、神经网络、数据分析。
本文借鉴了作者最近为《R语言数据分析挖掘必知必会 》课堂做的准备。
非常感谢您阅读本文,如需帮助请联系我们!


 2026脑机接口行业深度报告:市场规模、产业链、临床进展、政策赋能|附50+份报告PDF、数据、可视化模板汇总下载
2026脑机接口行业深度报告:市场规模、产业链、临床进展、政策赋能|附50+份报告PDF、数据、可视化模板汇总下载 2026年智旅新纪元AI旅游产业融合全景报告:智慧化、个性化、可持续|附240+份报告PDF、数据、可视化模板汇总下载
2026年智旅新纪元AI旅游产业融合全景报告:智慧化、个性化、可持续|附240+份报告PDF、数据、可视化模板汇总下载 Python酒店预订数据:随机森林与逻辑回归模型ROC曲线可视化
Python酒店预订数据:随机森林与逻辑回归模型ROC曲线可视化



