冗余分析(redundancy analysis,RDA)是一种回归分析结合主成分分析的排序方法,也是多因变量(multiresponse)回归分析的拓展。
从概念上讲,RDA是因变量矩阵与解释变量之间多元多重线性回归的拟合值矩阵的PCA分析。
本报告对植物生态多样性做了数据分析。冗余分析(Redundancy Analysis,RDA)确实是一种回归分析结合主成分分析的排序方法,同时也是多因变量(multi-response)回归分析的拓展。其主要目的是通过线性回归分析来寻找能够最大程度解释响应变量矩阵变差的一系列解释变量的线性组合,也就是环境对于样本的影响。因此,RDA可以被视为被解释变量约束的排序。本报告旨在对植物生态多样性进行数据分析,以揭示其分布、变化趋势及影响因素。通过收集相关数据集,运用统计方法和可视化技术,对植物生态多样性进行了深入研究。
可下载资源
作者

冗余分析
首先,加载数据。
要加载数据,所有文件都必须在工作目录中。
ste <- read.csv("sr.csv")
ev <- read.csv("ev.csv")
as <- read.csv("as.csv")
我对数据做了一些修改。首先,我将 ev 数据的所有定量变量(即除地貌单元外的所有变量)与 as 数据组合成一个名为 enqut. 然后,我对数据进行了归一化, 允许非常不同单位的变量之间进行比较。最后,我在归一化的定量环境变量中添加了地貌单元列,创建数据框 era,用于冗余分析。
enqut<- cbind(ev\[,-5\],ap) enz <- scale ut <- env\[,5\] era<- data.frame
结构数据
我使用环境数据era 作为解释变量对植被结构进行了冗余分析。我将结果分配给对象 str。
summary(str)
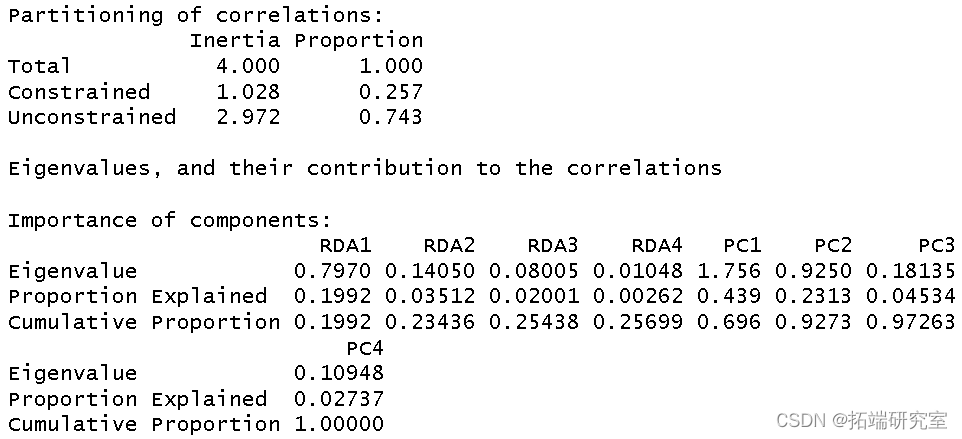
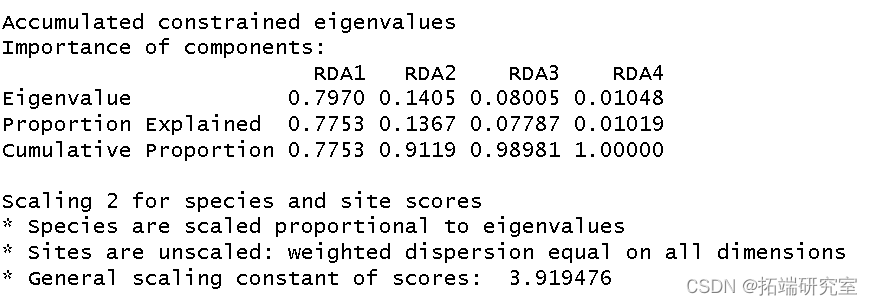
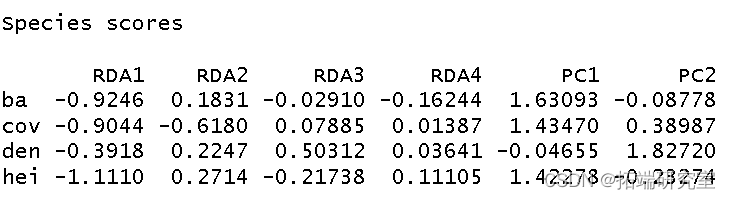
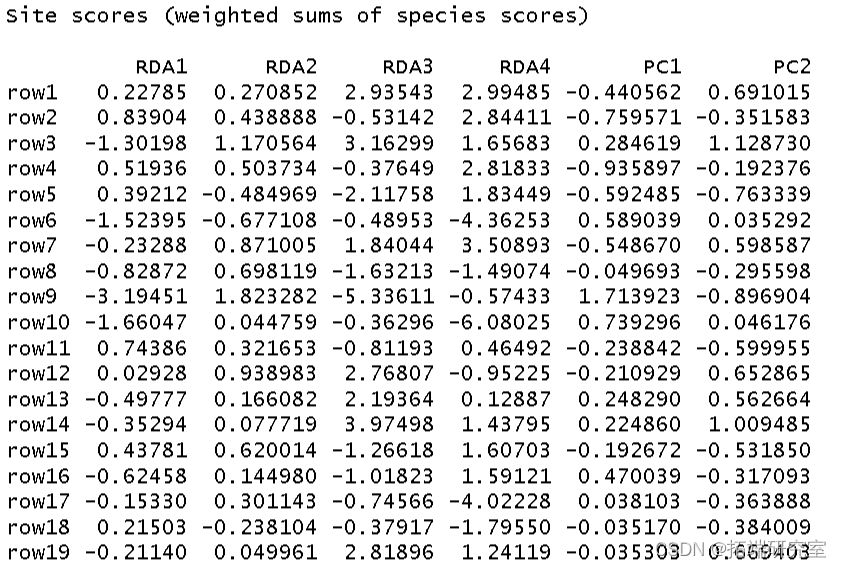
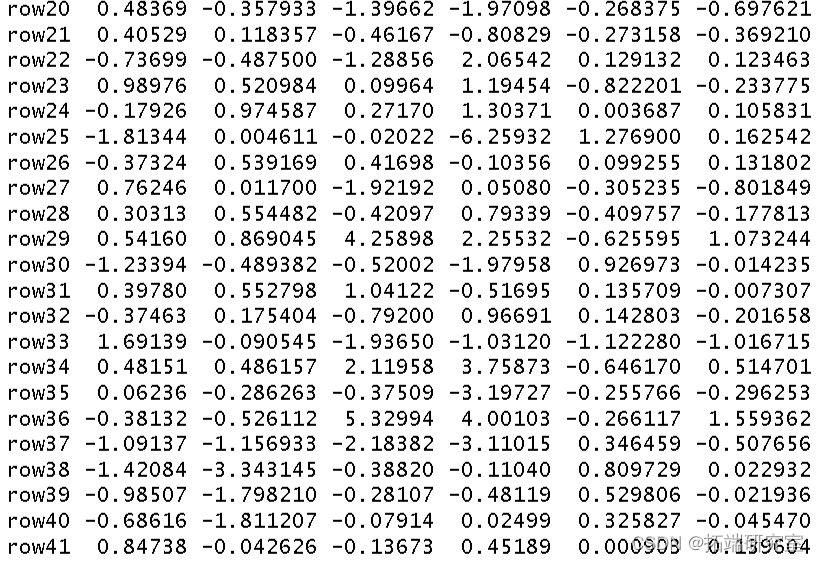
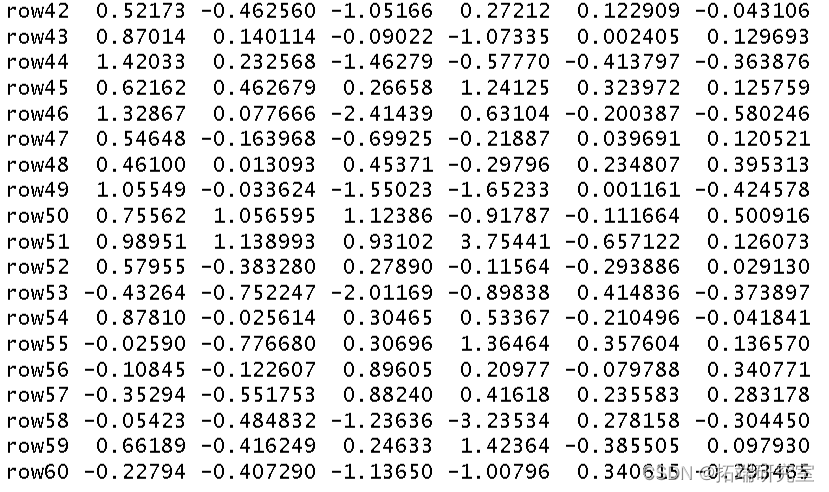
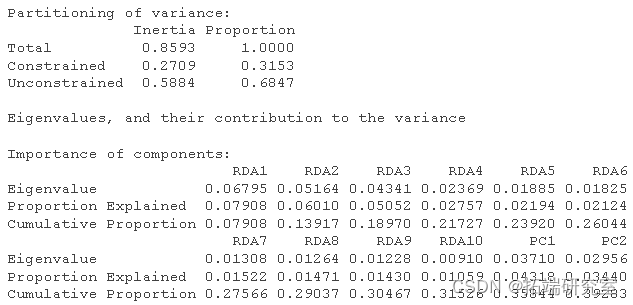
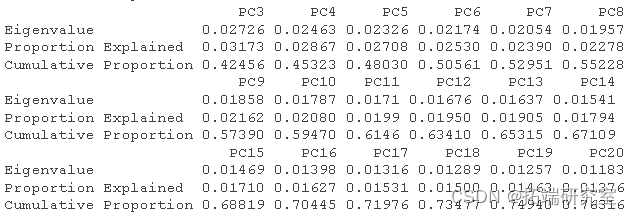
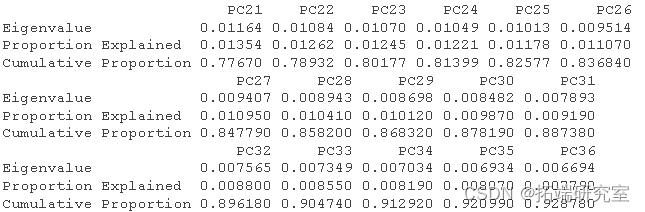
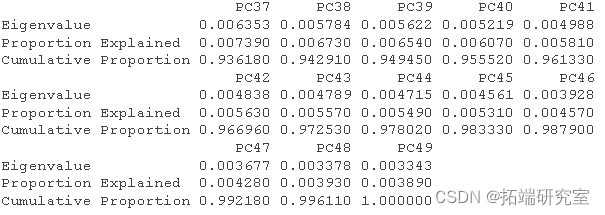
然后我得到了这个分析的 R 方和调整后R 方。
RsquareAdj
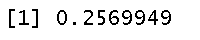
RsqeAdj$adj.r.sqd

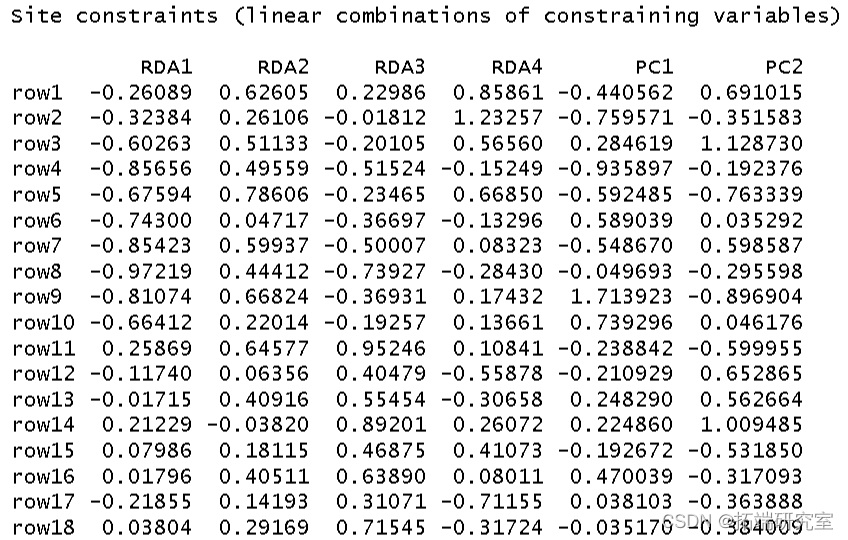
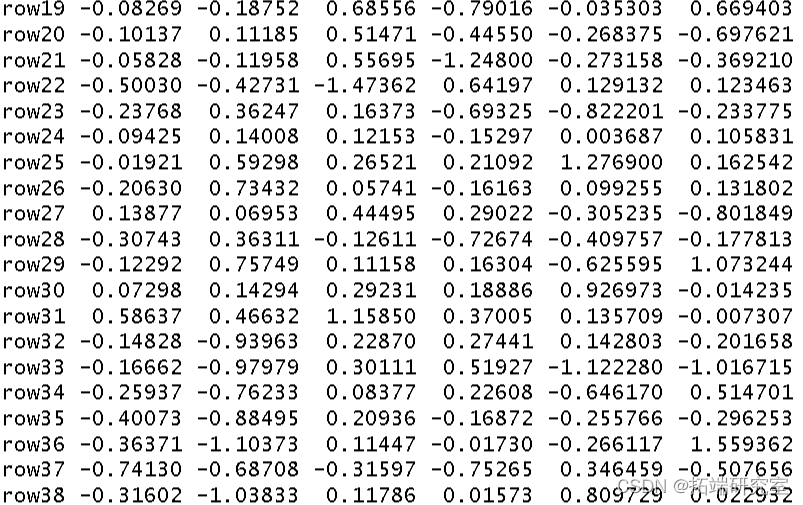
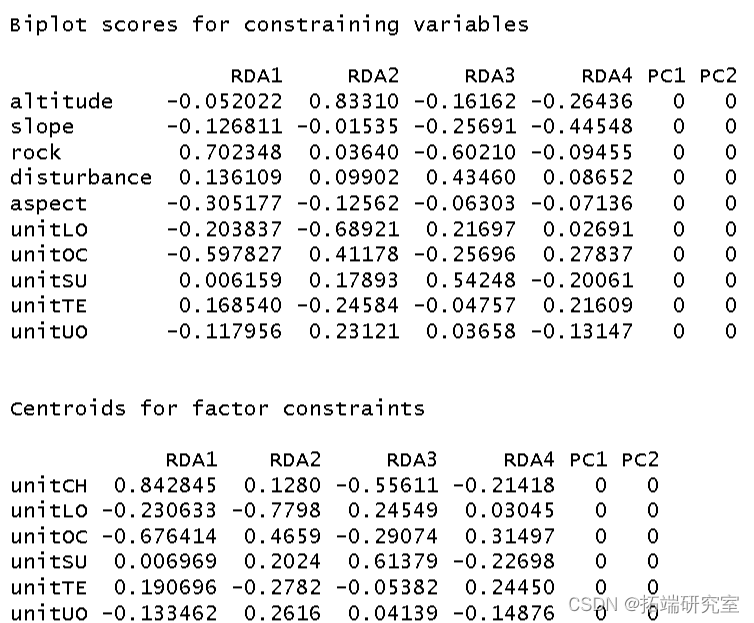
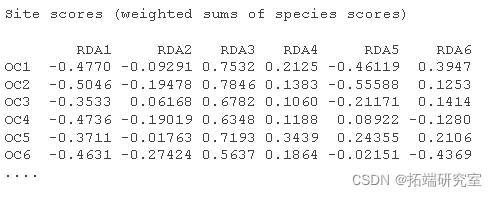
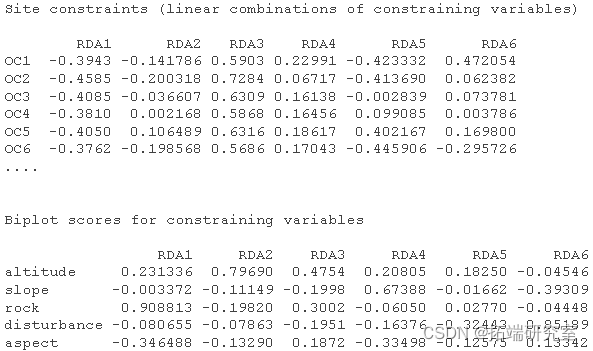
制作三序图。
par plot points usc <- scores points text
成分数据
首先我加载了物种数据。同样,该文件 PAl.csv 必须在工作目录中。为了降低大丰度的重要性,我将 Hellinger 转换应用于物种数据。
sp <- Hellinger(sp)
然后我使用所有环境变量作为解释变量进行了冗余分析。
head(suda)
随时关注您喜欢的主题
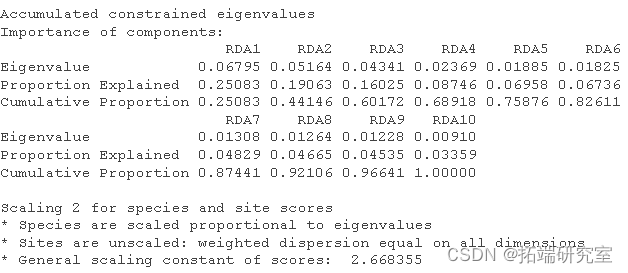

# 获得R^2和调整后的R^2 (sR2 <- RseAdj
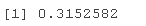
(spdj <- RseAdj$adj.r.sed)
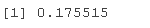
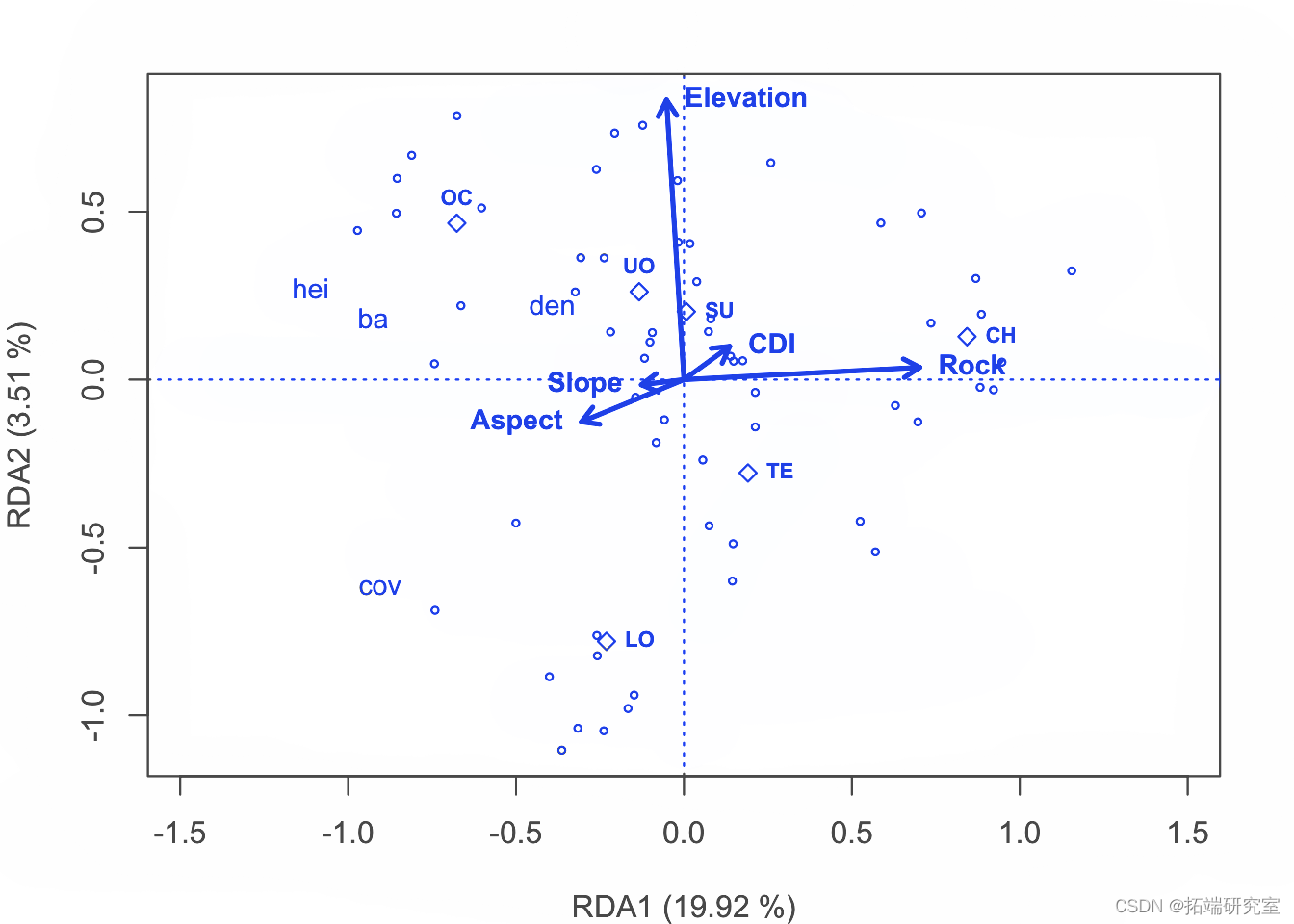
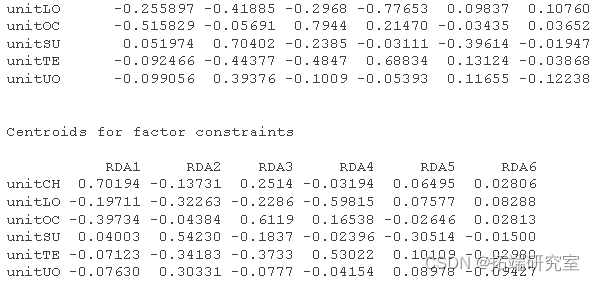
以2型标尺 对物种数据制作 RDA三序图。
# 做好绘图空间 par plot # 绘制站点的分数 spc <- scores points # 绘制出物种的点数 ssc <- scores points # 绘制定量解释变量的箭头和它们的标签 spesc <- scores arrows env.names text # 绘制地貌单元中心点和它们的标签的绘图点 spsc <- scores points text
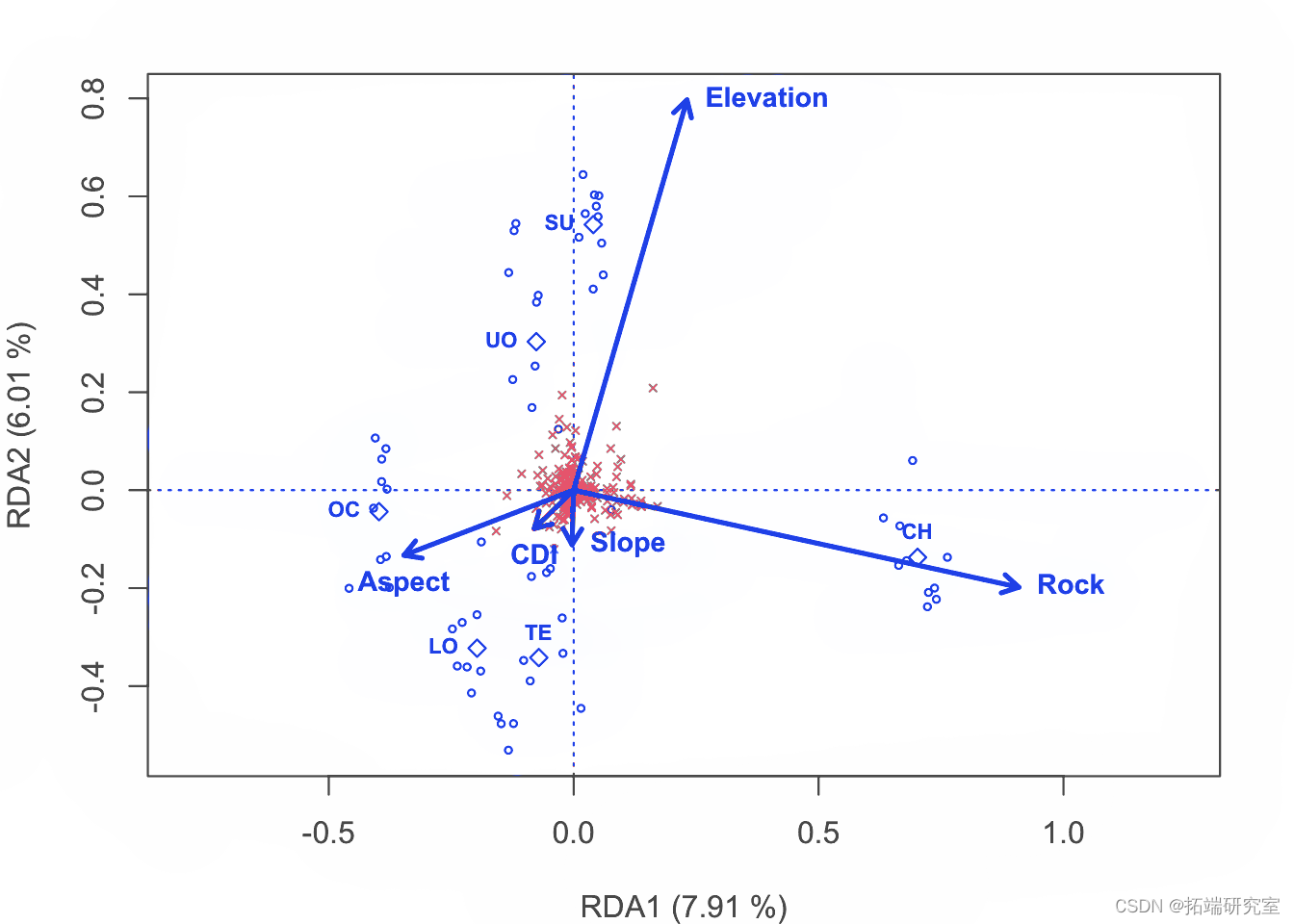
这是为论文制作图形的代码。
论文图形
par ensc <- scores arrows points # 制作绘图空间 par plot abline mtext # 绘制站点的分数 spsc <- scores points
# 绘制出物种的点数
sp.sc <- scores
points
# 绘制定量解释变量的箭头和它们的标签
spsc <- scores
arrows
text
# 绘制地貌单元中心点和它们的标签的绘图点
unimes
spusc <- scores
points
text
可下载资源
关于作者
Kaizong Ye是拓端研究室(TRL)的研究员。在此对他对本文所作的贡献表示诚挚感谢,他在上海财经大学完成了统计学专业的硕士学位,专注人工智能领域。擅长Python.Matlab仿真、视觉处理、神经网络、数据分析。
本文借鉴了作者最近为《R语言数据分析挖掘必知必会 》课堂做的准备。
非常感谢您阅读本文,如需帮助请联系我们!


 R软件线性模型与lmer混合效应模型对生态学龙类智力测试数据层级结构应用
R软件线性模型与lmer混合效应模型对生态学龙类智力测试数据层级结构应用 Python梯度提升模型GBM生态学研究:SFS、RandomizedSearchCV预测黑腿蜱种群分布丰度可视化
Python梯度提升模型GBM生态学研究:SFS、RandomizedSearchCV预测黑腿蜱种群分布丰度可视化 R语言广义线性混合模型GLMMs在生态学中应用可视化2实例合集|附数据代码
R语言广义线性混合模型GLMMs在生态学中应用可视化2实例合集|附数据代码 R语言非线性方程数值分析生物降解、植物生长数据:多项式、渐近回归、负指数方程、幂函数曲线、米氏方程、逻辑曲线、Gompertz、Weibull曲线
R语言非线性方程数值分析生物降解、植物生长数据:多项式、渐近回归、负指数方程、幂函数曲线、米氏方程、逻辑曲线、Gompertz、Weibull曲线
