
在当我们缺少值时,系统会告诉我用-1代替,然后添加一个指示符,该变量等于-1。这样就可以不删除变量或观测值。
我们在这里模拟数据,然后根据模型生成数据。未定义将转换为NA。一般建议是将缺失值替换为-1,然后拟合未定义的模型。默认情况下,R的策略是删除缺失值。如果未定义50%,则缺少数据,将删除一半的行
可下载资源
n=1000
x1=runif(n)
x2=runif(n)
e=rnorm(n,.2)
y=1+2*x1-x2+e
alpha=.05
indice=sample(1:n,size=round(n*alpha))
base=data.frame(y=y,x1=x1)
base$x1[indice]=NA
reg=lm(y~x1+x2,data=base)我们模拟10,000,然后看看未定义的分布,
m=10000
B=rep(NA,m)
hist(B,probability=TRUE,col=rgb(0,0,1,.4),border="white",xlab="missing values = 50%")
lines(density(B),lwd=2,col="blue")
abline(v=2,lty=2,col="red")视频
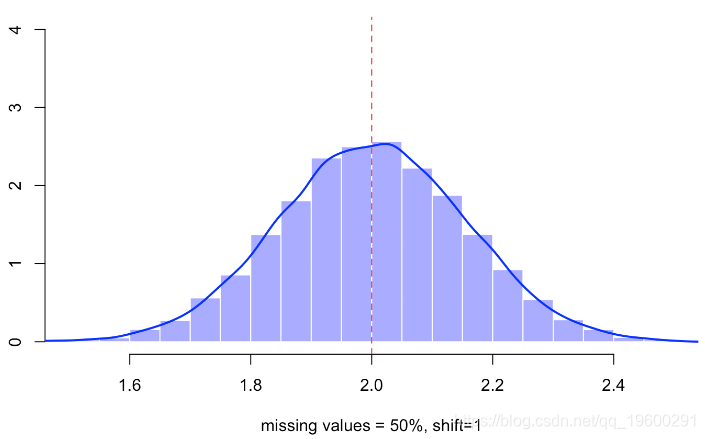
当然,丢失值的比率较低-丢失的观测值较少,因此估计量的方差较小。
现在让我们尝试以下策略:用固定的数值替换缺失的值,并添加一个指标,
B=rep(NA,m)
hist(B,probability=TRUE,col=rgb(0,0,1,.4),border="white")
lines(density(B),lwd=2,col="blue")
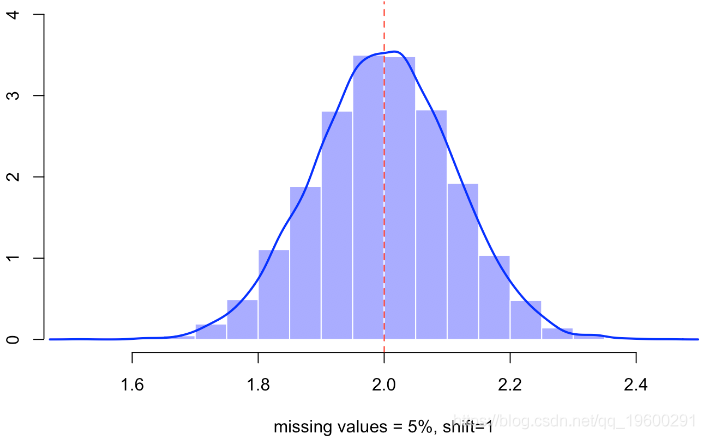
abline(v=2,lty=2,col="red")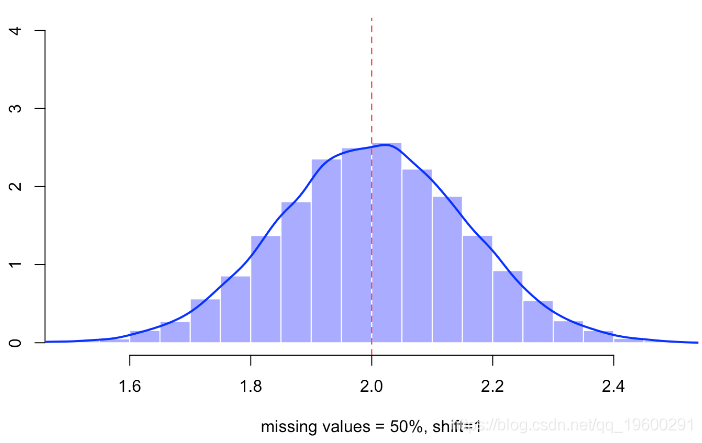
不会有太大变化,遗漏值的比率下降到5%,
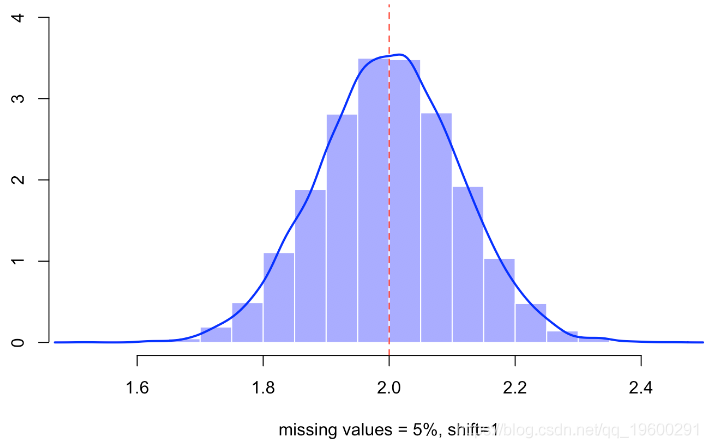
例如仍有5%的缺失值,我们有
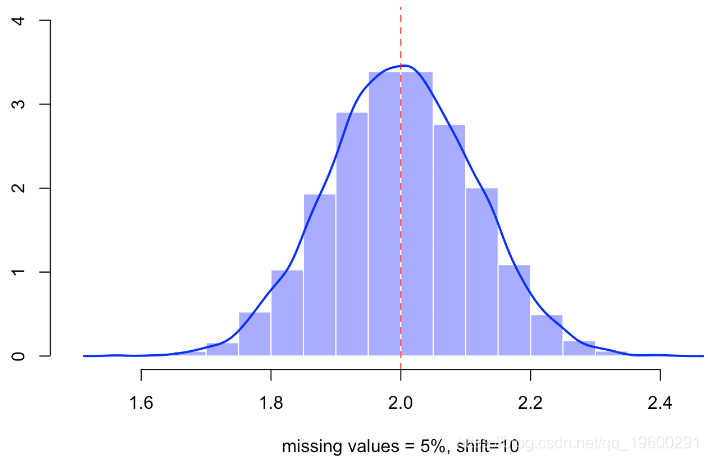
如果我们查看样本,尤其是未定义的点,则会观察到
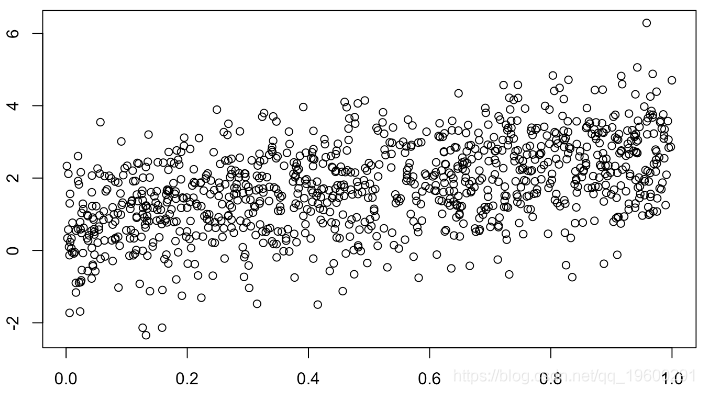
缺失值是完全独立地随机选择的,
x1=runif(n)
plot(x1,y,col=clr)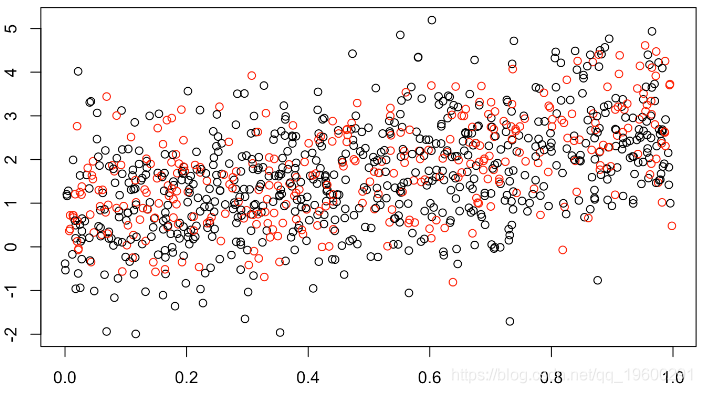
(此处缺失值的1/3为红色)。但可以假设缺失值的最大值,例如,
x1=runif(n)
clr=rep("black",n)
clr[indice]="red"
plot(x1,y,col=clr)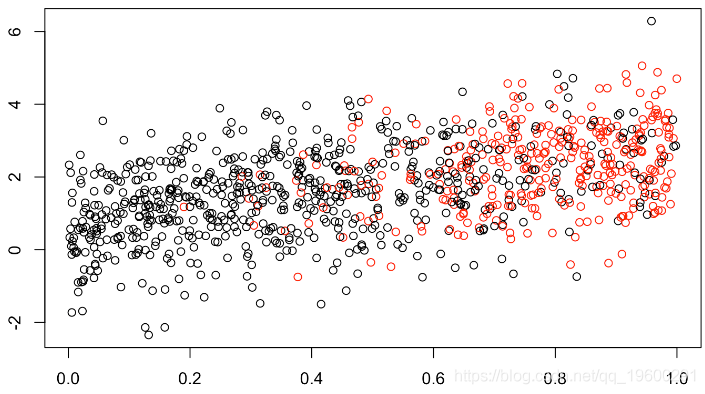
有人可能想知道,估计量会给出什么?
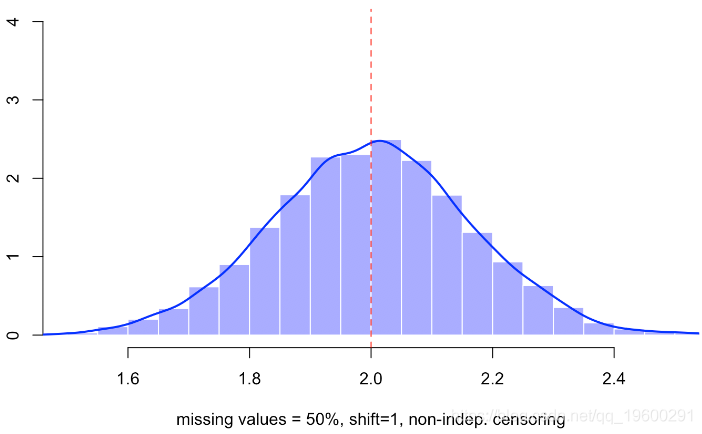
它变化不大,但是如果仔细观察,我们会有更多差异。如果未定义变量会发生什么,
for(s in 1:m){
base$x1[indice]=-1
reg=lm(y~x1+x2+I(x1==(-1)),data=base)
B[s]=coefficients(reg)[2]
}
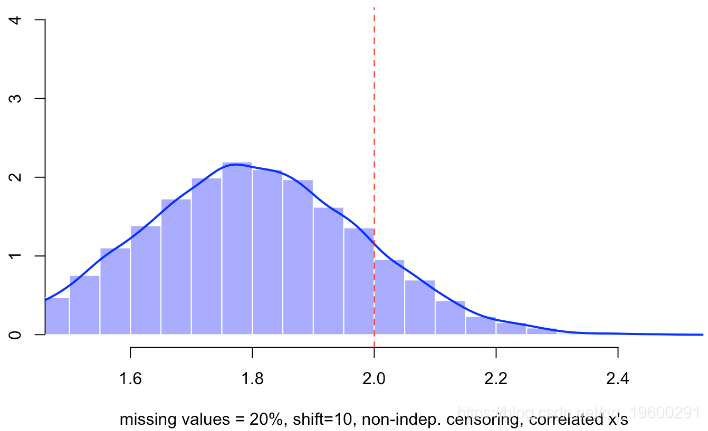
这次,我们有一个有偏差的估计量。
set.seed(1)
indice=sample(1:n,size=round(n*alpha),prob = x1^3)
base$x1[indice]=-1
coefficients(reg1)
(Intercept) x1 x2 I(x1 == (-1))TRUE
1.0988005 1.7454385 -0.5149477 3.1000668
base$x1[indice]=NA
coefficients(reg2)
(Intercept) x1 x2
1.1123953 1.8612882 -0.6548206正如我所说的,一种更好的方法是推算。这个想法是为未定义的缺失预测值预测。最简单的方法是创建一个线性模型,并根据非缺失值进行校准。然后在此新基础上估算模型。
for(s in 1:m){
base$x1[indice]=NA
reg0=lm(x1~x2,data=base[-indice,])
base$x1[indice]=predict(reg0,newdata=base[indice,])
reg=lm(y~x1+x2,data=base)
}
hist(B,probability=TRUE,col=rgb(0,0,1,.4),border="white")
lines(density(B),lwd=2,col="blue")
abline(v=2,lty=2,col="red")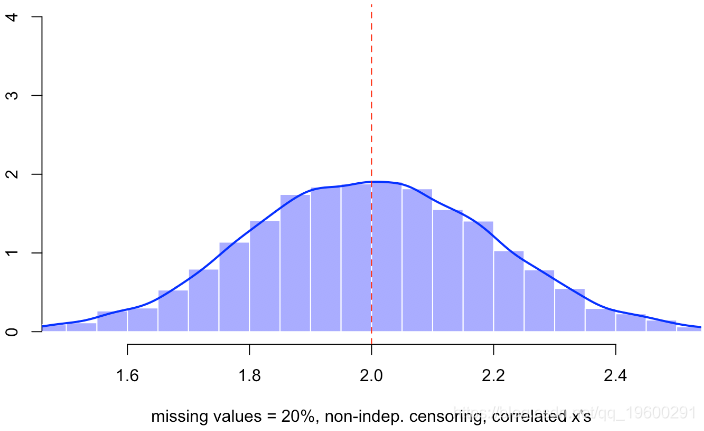
在数字示例中,我们得到
base$x1[indice]=NA
coefficients(reg3)
(Intercept) x1 x2
1.1593298 1.8612882 -0.6320339这种方法至少能够纠正偏差
然后,如果仔细观察,我们获得与第一种方法完全相同的值,该方法包括删除缺少值的行。
Coefficients:
Estimate Std. Error t value Pr(>|t|)
(Intercept) 1.15933 0.06649 17.435 < 2e-16 ***
x1 1.86129 0.21967 8.473 < 2e-16 ***
x2 -0.63203 0.20148 -3.137 0.00176 **
---
Signif. codes: 0 ‘***’ 0.001 ‘**’ 0.01 ‘*’ 0.05 ‘.’ 0.1 ‘ ’ 1
Residual standard error: 1.051 on 997 degrees of freedom
Multiple R-squared: 0.1094, Adjusted R-squared: 0.1076
F-statistic: 61.23 on 2 and 997 DF, p-value: < 2.2e-16
Coefficients: Estimate Std. Error t value Pr(>|t|)
(Intercept) 1.11240 0.06878 16.173 < 2e-16 ***
x1 1.86129 0.21666 8.591 < 2e-16 ***
x2 -0.65482 0.20820 -3.145 0.00172 **
---
Signif. codes: 0 ‘***’ 0.001 ‘**’ 0.01 ‘*’ 0.05 ‘.’ 0.1 ‘ ’ 1
Residual standard error: 1.037 on 797 degrees of freedom
(200 observations deleted due to missingness)
Multiple R-squared: 0.1223, Adjusted R-squared: 0.12
F-statistic: 55.5 on 2 and 797 DF, p-value: < 2.2e-16除了进行线性回归外,还可以使用另一种插补方法。
在模拟的基础上,我们获得
for(j in indice) base0$x1[j]=kpp(j,base0,k=5)
reg4=lm(y~x1+x2,data=base)
coefficients(reg4)
(Intercept) x1 x2
1.197944 1.804220 -0.806766如果我们看一下10,000个模拟中的样子,就会发现
for(s in 1:m){
base0=base
for(j in indice) base0$x1[j]=kpp(j,base0,k=5)
reg=lm(y~x1+x2,data=base0)
B[s]=coefficients(reg)[2]
}
hist(B,probability=TRUE,col=rgb(0,0,1,.4),border="white")
lines(density(B),lwd=2,col="blue")
abline(v=2,lty=2,col="red")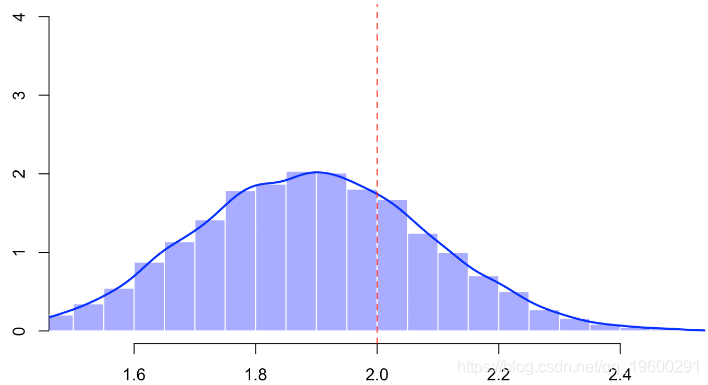
这里的偏差似乎比没有插补时要弱一些,换句话说,在我看来,插补方法似乎比旨在用任意值替换NA并在回归中添加指标的策略更强大。
可下载资源
关于作者
Kaizong Ye是拓端研究室(TRL)的研究员。在此对他对本文所作的贡献表示诚挚感谢,他在上海财经大学完成了统计学专业的硕士学位,专注人工智能领域。擅长Python.Matlab仿真、视觉处理、神经网络、数据分析。
本文借鉴了作者最近为《R语言数据分析挖掘必知必会 》课堂做的准备。
非常感谢您阅读本文,如需帮助请联系我们!

 R语言广义加性模型GAM、Tweedie分布的SaaS客户生命周期价值CLV预测研究——非线性关系捕捉与异方差性适配创新|附代码数据
R语言广义加性模型GAM、Tweedie分布的SaaS客户生命周期价值CLV预测研究——非线性关系捕捉与异方差性适配创新|附代码数据 R语言优化沪深股票投资组合:粒子群优化算法PSO、重要性采样、均值-方差模型、梯度下降法|附代码数据
R语言优化沪深股票投资组合:粒子群优化算法PSO、重要性采样、均值-方差模型、梯度下降法|附代码数据 视频讲解|Stata和R语言自助法Bootstrap结合GARCH对sp500收益率数据分析
视频讲解|Stata和R语言自助法Bootstrap结合GARCH对sp500收益率数据分析



