对“NCI60”(癌细胞系微阵列)数据使用聚类方法
目的是找出观察结果是否聚类为不同类型的癌症。
K_means 和层次聚类的比较。
可下载资源
#数据信息 dim(nata)
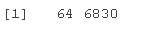
nci.labs\[1:4\]
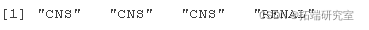
table(ncibs)
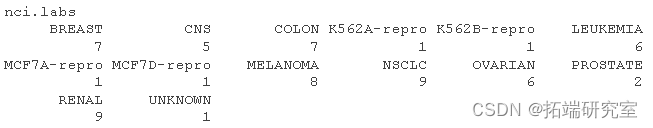
ncbs

scale # 标准化变量(均值零和标准差一)。
全链接、平均链接和单链接之间的比较。
plot(hclust,ylab = "",cex=".5",col="blue") #使用全链接对观察结果进行层次聚类。

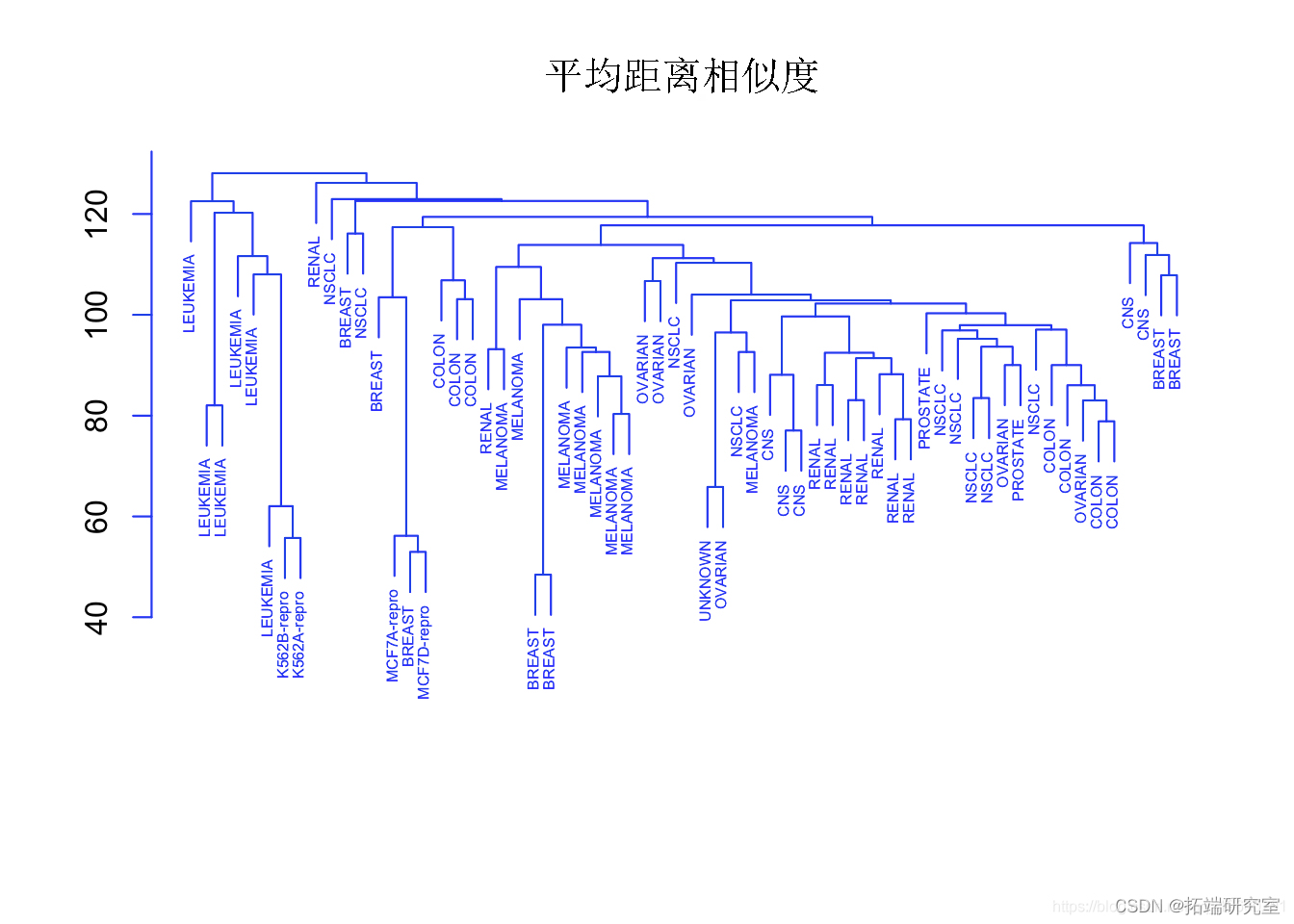
plot(hclust,cex=".5",col="blue") #使用平均链接对观察进行层次聚类。
par(mfrow=c(1,1)) plot(hclust,col="blue") #使用单链接对观察进行层次聚类。

观察结果
单链接聚类倾向于产生拖尾的聚类:非常大的聚类,单个观测值一个接一个地附在其中。
另一方面,全链接和平均链接往往会产生更加平衡和有吸引力的聚类。
由于这个原因,全链接和平均链接比单链接层次聚类更受欢迎。单一癌症类型中的细胞系确实倾向于聚在一起,尽管聚类并不完美。
随时关注您喜欢的主题
table(hrs,ncbs)

我们可以看到一个清晰的模式,即所有白血病细胞系都属于聚类 3,其中乳腺癌细胞分布在三个不同的聚类中。
plot(hcu) abline
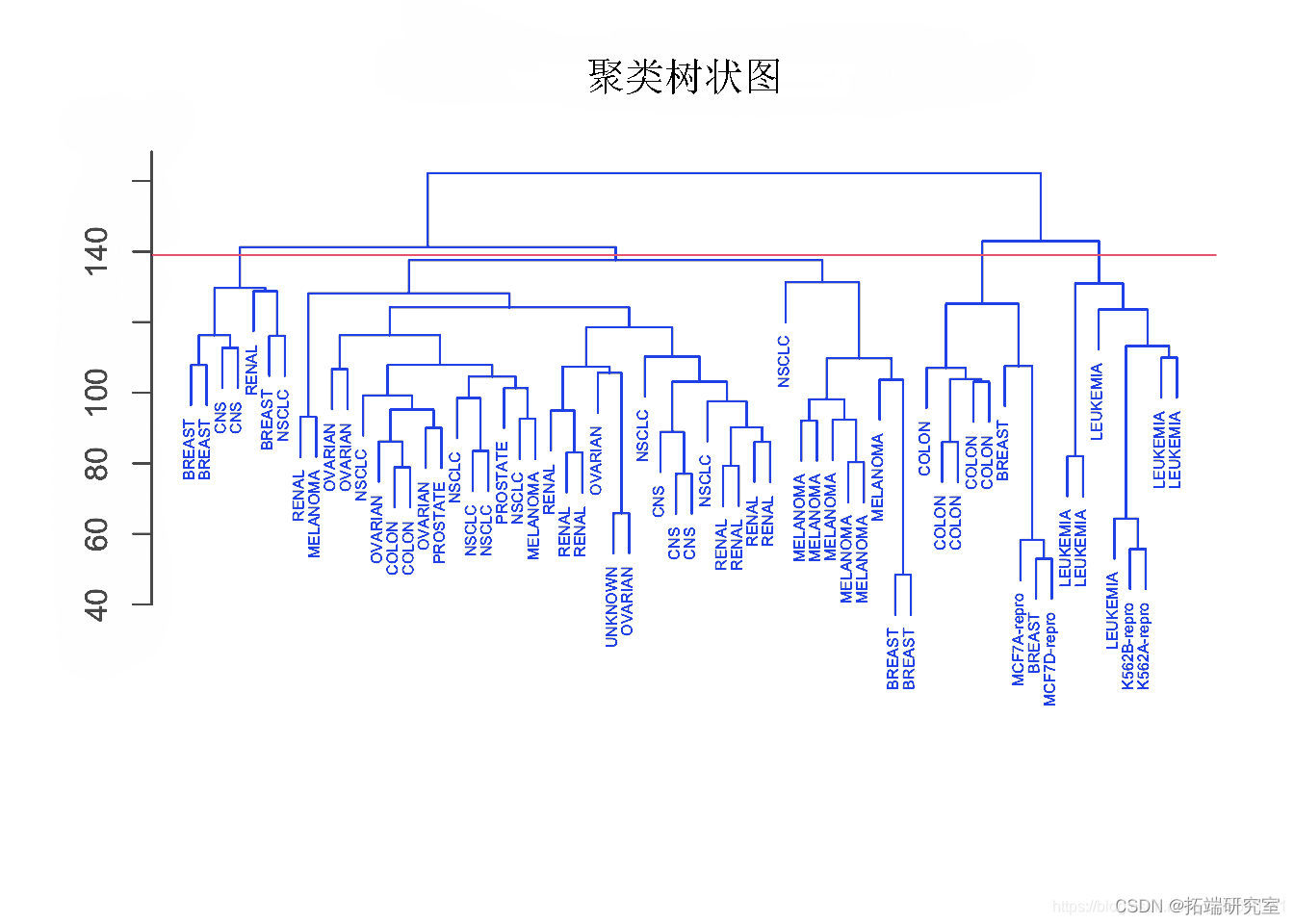
参数 h=139 在高度 139 处绘制一条水平线。这是 4 个不同聚类的划分结果。
out

kout=kmea table

我们看到,获得层次聚类和 K-means 聚类的四个聚类产生了不同的结果。K-means 聚类中的簇 2 与层次聚类中的簇 3 相同。另一方面,其他集群不同。
结论
层次聚类在 NCI60 数据集中能比 K-means聚类得到更好的聚类。
可下载资源
关于作者
Kaizong Ye是拓端研究室(TRL)的研究员。在此对他对本文所作的贡献表示诚挚感谢,他在上海财经大学完成了统计学专业的硕士学位,专注人工智能领域。擅长Python.Matlab仿真、视觉处理、神经网络、数据分析。
本文借鉴了作者最近为《R语言数据分析挖掘必知必会 》课堂做的准备。
非常感谢您阅读本文,如需帮助请联系我们!


 Python与R语言用XGBOOST、NLTK、LASSO、决策树、聚类分析电商平台评论文本信息数据集
Python与R语言用XGBOOST、NLTK、LASSO、决策树、聚类分析电商平台评论文本信息数据集 Python对Airbnb北京、上海链家租房数据用逻辑回归LR、决策树、岭回归、Lasso、随机森林、XGBoost、神经网络kmeans聚类分析市场影响因素|数据分享
Python对Airbnb北京、上海链家租房数据用逻辑回归LR、决策树、岭回归、Lasso、随机森林、XGBoost、神经网络kmeans聚类分析市场影响因素|数据分享 专题|R语言、SPSS电信客户流失预测实例汇总:KNN、决策树、聚类、RFM分群、挽留策略研究
专题|R语言、SPSS电信客户流失预测实例汇总:KNN、决策树、聚类、RFM分群、挽留策略研究 Python用K-Means聚类、 LRFMC模型对航空公司客户数据价值可视化分析指标应用
Python用K-Means聚类、 LRFMC模型对航空公司客户数据价值可视化分析指标应用


