
本文聚焦于图注意力网络GAT在蛋白质 – 蛋白质相互作用数据集中的应用。
首先介绍了研究背景与目的,阐述了相关概念如归纳设置与转导设置的差异。
接着详细描述了数据加载与可视化的过程,包括代码实现与分析,如 数据集的读取、处理以及图数据加载器的构建等。
通过对数据形状和类型的分析,深入理解数据特性。最后强调了在项目开发过程中测试代码以及可视化的重要性,为 GAT 在 数据集上的进一步研究与应用奠定基础。
一、引言
本研究围绕图注意力网络(GAT)展开,重点探讨其在蛋白质相互作用数据集中的应用。GAT 作为一种强大的图神经网络模型,在处理具有复杂结构的数据时展现出独特的优势。本文本旨在解释如何在归纳设置下使用 GAT,并以 数据集为例进行深入研究。通过对 蛋白质 数据集的分析与处理,期望能够为生物信息学等领域的研究提供有力的技术支持与理论依据。
二、相关概念
(一)归纳设置与转导设置
在图神经网络中,归纳设置和转导设置是两种不同的数据处理方式。转导设置通常针对单个图,例如 Cora 数据集,将图中的一些节点(而非图本身)划分为训练、验证和测试集。
在训练过程中,仅使用训练节点的标签信息,但在正向传播时,由于空间 GNN 的工作原理,会聚合邻居节点的特征向量,其中部分邻居节点可能属于验证集甚至测试集。这里主要利用了邻居节点的结构信息和特征,而非其标签信息。
而归纳设置则更类似于计算机视觉或自然语言处理中的常见方式。在这种设置下,拥有一组训练图、一组独立的验证图和一组独立的测试图。这种设置使得模型能够在不同的图数据上进行训练和评估,具有更强的泛化能力。
三、数据加载与可视化
(一)数据加载
在数据加载部分,首先定义了一些必要的函数和类。例如,json_read 函数用于读取 JSON 格式的数据:
def json_read(path):
with open(path, 'r') as file:
data = json.load(file)
return data
该函数接受一个文件路径作为参数,打开文件并读取其中的 JSON 数据,最后返回读取的数据。load_grata 函数则用于加载 蛋白质 数据集的图数据:
视频
Python、R时间卷积神经网络TCN与CNN、RNN预测时间序列实例
视频
【视频讲解】Python深度学习股价预测、量化交易策略:LSTM、GRU深度门控循环神经网络附代码数据
视频
【视频讲解】神经网络、Lasso回归、线性回归、随机森林、ARIMA股票价格时间序列预测
视频
LSTM神经网络架构和原理及其在Python中的预测应用
视频
卷积神经网络CNN肿瘤图像识别
if dataset_name == DatasetType.蛋白质.name.lower(): # 蛋白质 - 蛋白质相互作用数据集
# 若 蛋白质 数据路径不存在,则首次使用时下载
if not os.path.exists(蛋白质_PATH):
os.makedirs(蛋白质_PATH)
# 步骤 1:下载 蛋白质.zip(包含 蛋白质 数据集)
zip\\\_tmp\\\_path = os.path.join(蛋白质_PATH, '蛋白质.zip')
download\\\_url\\\_to\\\_file(蛋白质\\\_URL, zip\\\_tmp\\\_path)
# 步骤 2:解压
with zipfile.ZipFile(zip\\\_tmp\\\_path) as zf:
zf.extractall(path=蛋白质_PATH)
print(f'解压至: {蛋白质_PATH} 完成。')
# 步骤 3:删除临时资源文件该函数根据配置信息加载 蛋白质 数据集,包括下载数据(若不存在)、读取节点特征、标签和图拓扑结构等,并将数据整理为适合训练的格式,最后返回相应的数据加载器。
GraphD 类用于从分割中获取单个图数据:
class GraphDt(Dataset):
def \\_\\\_init\\\_\\_(self, node\\\_features\\\_list, node\\\_labels\\\_list, edge\\\_index\\\_list):
self.node\\\_features\\\_list = node\\\_features\\\_list
self.node\\\_labels\\\_list = node\\\_labels\\\_list
self.edge\\\_index\\\_list = edge\\\_index\\\_list
# 需定义 len 和 getitem 函数以便 DataLoader 正常工作
def \\_\\\_len\\\_\\_(self):
return len(self.edge\\\_index\\\_list)
(二)数据可视化
为了可视化数据:
if should_visualize:
plot\\\_in\\\_out\\\_degree\\\_distributions(edge\\\_index.numpy(), graph.number\\\_of\\\_nodes(), dataset\\\_name)
visualize\\\_graph(edge\\\_index.numpy(), node\\\_labels\\\[mask\\\], dataset\\\_name)
四、数据形状与类型分析
通过加载数据并获取一批训练数据,对数据的形状和类型进行了分析。以特定的 蛋白质 训练图(批次大小为 1)为例,其具有 3021 个节点,每个节点有 50 个特征,这与 蛋白质 数据集的特性相关,每个节点的特征是多种基因集信息的组合。蛋白质 数据集共有 121 个类别,且每个节点可以关联多个类别,属于多标签分类数据集。该图包含 94359 条边(包括自环),与 Cora 数据集的 13k 条边相比数量较多。此外,边索引的数据类型为 int64,这是由于 PyTorch 中 index_select 函数的要求,而节点标签可以使用 float32 类型,因为 nn.BtsLoss 不需要 long/int64 类型,这样可以节省内存。
基于蛋白质相互作用网络的数据可视化与图注意力网络(GAT)模型研究
接下来聚焦于蛋白质相互作用网络,深入探讨其数据可视化与图注意力网络(GAT)模型的应用。通过详细分析节点度分布、构建并训练 GAT 模型以及对模型进行可视化分析,揭示了 蛋白质 网络的结构特征与 GAT 模型在多标签分类任务中的有效性,为相关领域的研究提供了有价值的参考。
一、
在生物信息学领域,蛋白质相互作用网络的研究具有至关重要的意义。理解 蛋白质 网络的结构和特性,有助于深入探究蛋白质的功能以及生物体内的复杂生理过程。本文旨在通过数据可视化和构建图注意力网络(GAT)模型,对 蛋白质 网络进行全面的分析与研究,为相关领域的进一步探索奠定基础。
二、蛋白质 数据可视化
(一)节点度分布可视化
为了初步了解 蛋白质 网络中节点的连接情况,我们首先研究节点的度分布,即节点拥有的输入/输出边的数量,这是衡量图连通性的一个重要指标。
运行以下代码以可视化 蛋白质 的度分布:
num\\\_of\\\_nodes = len(node_labels)
plot\\\_in\\\_out\\\_degree\\\_distributions(edge\\\_index, num\\\_of\\\_nodes, config\\\['dataset\\\_name'\\\])

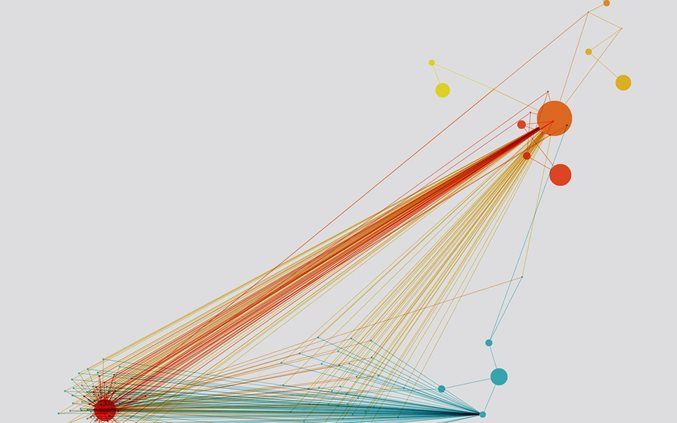
(二)蛋白质 图可视化
接下来,我们将可视化 蛋白质 图。以下代码用于构建和绘制 蛋白质 图:
dataset\\\_name = config\\\['dataset\\\_name'\\\]
visualization_tool = GraphVisualizationTool.IGRAPH
# 如果 edge_index 是 torch.Tensor 类型,则将其转换为 numpy 数组
if isinstance(edge_index, torch.Tensor):
edge\\\_index\\\_np = edge_index.cpu().numpy()
# 如果 node_labels 是 torch.Tensor 类型,则将其转换为 numpy 数组
if isinstance(node_labels, torch.Tensor):

从可视化结果可以得出以下结论:
- 由于我们将 蛋白质 视为无向图,因此前两个图相同。
- 与 Cora 相比,更多的节点具有大量的边,但大多数节点的边数仍然较少。
- 第三个图以直方图的形式清晰地展示了这一点,大多数节点只有 1 – 20 条边(因此在最左侧有峰值),并且与 Cora 相比,分布更为分散。
需要注意的是,我不得不清除此单元格的原始输出,否则文件会非常大。这里仅展示了一个任意的 蛋白质 训练图示例,结果可能会有所不同(共有 20 个训练图)。
GAT 模型理解
GAT 模型类定义
首先创建一个高级类,用于构建 GAT 模型。该类主要将各层堆叠到对象中,并将数据(特征、边索引)打包成元组。
class GAT(torch.nn.Module):
"""
最有趣且最具挑战性的实现是实现 #3。
Imp1 和 imp2 在细节上有所不同,但基本相同。
因此,在本笔记本中,我将重点关注 imp #3。
"""
def \\_\\\_init\\\_\\_(self, num\\\_of\\\_layers, num\\\_heads\\\_per\\\_layer, num\\\GAT 层定义
接下来定义 GATLayer 类,该类是 GAT 模型的核心组成部分。
但希望它更具可读性!(并且性能相似)
"""
# 源节点在边索引中的维度位置
src\\\_nodes\\\_dim = 0
# 目标节点在边索引中的维度位置
trg\\\_nodes\\\_dim = 1
# 节点维度(在张量中 "N" 的位置,axis 可能是更熟悉的术语)
nodes_dim = 0
# 注意力头维度
head_dim = 1
def \\_\\\_init\\\_\\_(self, num\\\_in\\\_features, num\\\_out\\\_features, num\\\_of\\\_heads, concat=True, activation=nn.ELU(),
dropout\\\_prob=0.6, add\\\_skip\\\_connection=True, bias=True, log\\\_attention_weights=False):
super().\\_\\\_init\\\_\\_()
self.num\\\_of\\\_heads = num\\\_of\\\_heads
训练 GAT 模型(蛋白质 多标签分类)
相关常量定义
随时关注您喜欢的主题
首先定义一些训练相关的常量,包括训练阶段枚举、日志记录器、早停相关变量以及模型保存路径等。
from torch.utils.tensorboard import SummaryWriter
# 3 种不同的模型训练/评估阶段,用于 train.py
class LoopPhase(enum.Enum):
TRAIN = 0,
基于图注意力网络(GAT)的模型训练与可视化分析
接下来我们详细阐述了图注意力网络(GAT)在特定数据集(如 蛋白质)上的训练过程及相关可视化分析。通过定义一系列实用函数来构建训练模型所需的组件,包括数据加载、模型架构定义、训练循环设置等,并对训练得到的模型进行注意力和熵可视化,以深入理解 GAT 模型的学习效果与特性。
一、引言
图注意力网络(GAT)在处理图结构数据方面具有重要意义。在本文中,我们将深入探讨其在 蛋白质 数据集上的应用,涵盖从模型训练到可视化分析的完整流程,旨在揭示 GAT 模型在该数据集上的表现及内在机制。
二、模型训练相关函数定义
(一)获取训练状态函数
import git
import re # 正则表达式模块
def get\\\_training\\\_state(training_config, model):
training_state = {
# 获取代码仓库的提交哈希值
"commit\\\_hash": git.Repo(search\\\_parent_directories=True).head.object.hexsha,
该函数用于收集训练过程中的重要信息,包括代码版本信息(通过提交哈希值体现)、训练数据集名称、训练轮数、测试性能指标以及模型的结构和参数状态等。
这些信息对于后续的模型分析、比较和复现具有重要价值。
(二)打印模型元数据函数
def print\\\_model\\\_metadata(training_state):
# 构建打印头部信息
header = f'\\\n{"*"\\\*5} Model training metadata: {"\\\*"*5}'
print(header)
for key, value in training_state.items():
# 不打印模型参数字典,因为其内容为大量数字
if key!= 'state_dict':
print(f'{key}: {value}')
print(f'{"*" * len(header)}\\\n')
此函数用于以清晰的格式打印模型训练的元数据,除了模型参数字典外,将其他关键信息如数据集名称、训练轮数等展示出来,方便用户快速了解模型训练的基本情况。
三、命令行参数解析函数
此函数利用 argparse 模块解析命令行参数,涵盖训练过程中的各种设置,如训练轮数、学习率、是否使用 GPU 等,同时也包括数据集相关和日志记录相关的参数。通过合理设置这些参数,可以灵活地调整模型训练过程,满足不同的实验需求。
四、GAT 模型训练函数
['force\\\_cpu'\\\] else "cpu")
# 步骤 1:准备数据加载器
data\\\_loader\\\_train, data\\\_loader\\\_val, data\\\_loader\\\_test = load\\\_graph\\\_data(config, device)
# 步骤 2:准备模型
gat = GAT(
num\\\_of\\\_layers=config\\\['num\\\_of\\\_layers'\\\],
num\\\_heads\\\_per\\\_layer=config\\\['num\\\_heads\\\_per\\\_layer'\\\],
num\\\_features\\\_per\\\_layer=config\\\['num\\\_features\\\_per\\\_layer'\\\],
该函数是 GAT 模型在 蛋白质 数据集上的训练主函数,按照特定的步骤进行操作。首先根据设备情况(GPU 或 CPU)准备数据加载器,然后构建 GAT 模型并定义损失函数和优化器,接着通过装饰器函数简化训练和验证循环,最后在训练过程中进行训练循环、验证循环,并根据需要进行测试,最终将训练得到的模型状态保存下来。
图注意力网络(GAT)的熵直方图可视化分析
摘要: 接下来聚焦于图注意力网络(GAT)中熵直方图的可视化研究。阐述了熵概念在 GAT 模型分析中的引入缘由,详细介绍了相关函数的构建与作用,包括绘制熵直方图函数以及整体可视化函数。通过在 蛋白质 数据集上的应用与结果展示,深入探讨了 GAT 模型学习到的注意力模式与均匀注意力模式的差异,为理解 GAT 模型的学习效果提供了重要视角。
熵直方图可视化原理
在 GAT 模型的研究中,熵直方图可视化是一种重要的分析手段。当提及“熵”时,人们可能会疑惑它在此处的作用。事实上,这并不复杂。在 GAT 模型中,注意力系数总和为 1,这就形成了一种概率分布。而有概率分布就可以计算熵,熵能够量化分布中的信息量(对于专业人士而言,它是自信息的期望值)。若对熵的概念不熟悉,可参考精彩视频,不过在理解本研究的可视化目的时,并不需要深入掌握熵的理论。
其核心思想如下:假设有一个“假设性的”GAT 模型,它对每个节点的邻域具有恒定的注意力(即所有分布是均匀的),我们计算每个邻域的熵,并根据这些熵值绘制直方图。然后将其与我们训练得到的 GAT 模型的直方图进行比较,观察两者的差异。如果两个直方图完全重叠,意味着我们的 GAT 模型具有均匀的注意力模式;重叠越小,则分布越不均匀。在此,我们关注的并非信息本身,而是直方图的匹配程度。这有助于清晰地了解 GAT 模型学习到的注意力模式是否有意义。若 GAT 学习到的是恒定注意力,那么使用 GCN 或更简单的模型可能就足够了。
实验运行与结果分析
最后运行 函数:
visualize\\\_entropy\\\_histograms(
model_name,
dataset_name,
)
得到的结果如以下图片所示:



从结果可以看出,浅蓝色直方图(训练后的 GAT)与橙色直方图(均匀注意力 GAT)相比发生了倾斜。并且由于均匀分布具有最高的熵,所以它们向左倾斜,这是符合预期的。如果之前通过边厚度绘制的可视化结果未能使您信服,那么熵直方图的结果将更具说服力。通过熵直方图可视化,我们能够更深入地理解 GAT 模型在 蛋白质 数据集上学习到的注意力模式与均匀注意力模式的差异,从而评估 GAT 模型的有效性和独特性,为进一步优化和应用 GAT 模型提供有力的依据。
每日分享最新报告和数据资料至会员群
关于会员群
- 会员群主要以数据研究、报告分享、数据工具讨论为主;
- 加入后免费阅读、下载相关数据内容,并同步海内外优质数据文档;
- 老用户可九折续费。
- 提供报告PDF代找服务
非常感谢您阅读本文,如需帮助请联系我们!



 SPSS与Python用Resblock优化BP神经网络分析慢性胃炎病历数据聚类K-means/AGNES、关联规则挖掘及预测
SPSS与Python用Resblock优化BP神经网络分析慢性胃炎病历数据聚类K-means/AGNES、关联规则挖掘及预测 Python神经网络、随机森林、PCA、SVM、KNN及回归实现ERα拮抗剂、ADMET数据预测|附代码数据
Python神经网络、随机森林、PCA、SVM、KNN及回归实现ERα拮抗剂、ADMET数据预测|附代码数据 Python农作物种植策略研究GA-BP神经网络、蒙特卡洛算法、自注意力Stacking集成模型及粒子群算法PSO优化基于华北山区乡村农作物数据及地块数据
Python农作物种植策略研究GA-BP神经网络、蒙特卡洛算法、自注意力Stacking集成模型及粒子群算法PSO优化基于华北山区乡村农作物数据及地块数据 MATLAB奥运会奖牌预测研究 —CNN神经网络、逻辑回归、Liang-Kleeman信息流、多元回归及随机森林模型的因果关联与概率预测|附代码数据
MATLAB奥运会奖牌预测研究 —CNN神经网络、逻辑回归、Liang-Kleeman信息流、多元回归及随机森林模型的因果关联与概率预测|附代码数据



