
r语言中如何进行两组独立样本秩和检验
所述配对双样品的Wilcoxon检验一种的非参数检验,其可以被用于比较样品的两个独立数据。
可下载资源
本文介绍如何在ř中计算两个样本的秩检验。
可视化数据并在R中计算的Wilcoxon测试
R函数用于计算的秩检验
为了执行两个样本的Wilcoxon检验,比较两个独立样本(x&y)的均值,R函数wilcox.test()可以如下使用:
wilcox.test(x, y, alternative = "two.sided")- x,y:数字向量
- 替代方案:替代假设允许值是“two.sided”(默认值),“更大”或“更少”之一。
将数据导入R
- 准备数据
- 将数据保存在外部的.TXT选项卡或的的.csv文件中
- 将您的数据导入ř如下:
#.txt 文件
my_data <- read.delim(file.choose())
# .csv文件
my_data <- read.csv(file.choose())在这里,我们将使用一个示例数据集,其中包含18个人(9名女性和9名男性)的权重:
#
women_weight <- c(38.9, 61.2, 73.3, 21.8, 63.4, 64.6, 48.4, 48.8, 48.5)
men_weight <- c(67.8, 60, 63.4, 76, 89.4, 73.3, 67.3, 61.3, 62.4)
# 创建数据框
my_data <- data.frame(
group = rep(c("Woman", "Man"), each = 9),
weight = c(women_weight, men_weight)
)我们想知道,如果女性体重的中位数与男性体重的中位数不同?
检查数据
print(my_data) group weight
1 Woman 38.9
2 Woman 61.2
3 Woman 73.3
4 Woman 21.8
5 Woman 63.4
6 Woman 64.6
7 Woman 48.4
8 Woman 48.8
9 Woman 48.5
10 Man 67.8
11 Man 60.0
12 Man 63.4
13 Man 76.0
14 Man 89.4
15 Man 73.3
16 Man 67.3
17 Man 61.3
18 Man 62.4可以按组计算汇总统计数据(中位数和四分位数间距(IQR))。可以使用dplyr包。
- 要安装dplyr软件包,请键入以下内容:
install.packages("dplyr")- 按组计算摘要统计信息:
library(dplyr)
group_by(my_data, group) %>%
summarise(
count = n(),
median = median(weight, na.rm = TRUE),
IQR = IQR(weight, na.rm = TRUE)
)Source: local data frame [2 x 4]
group count median IQR
(fctr) (int) (dbl) (dbl)
1 Man 9 67.3 10.9
2 Woman 9 48.8 15.0使用箱形图可视化数据
您可以按照此链接中的描述绘制R基本图:R基本图。在这里,我们将使用ggpubr R包进行基于ggplot2的简单数据可视化
- 从GitHub上的安装最新版本的ggpubr如下(推荐):
# 安装
if(!require(devtools)) install.packages("devtools")
devtools::install_github("kassambara/ggpubr")- 或者,从CRAN安装如下:
install.packages("ggpubr")- 可视化您的数据:
# 分组绘图
library("ggpubr")
ggboxplot(my_data, x = "group", y = "weight",
color = "group", palette = c("#00AFBB", "#E7B800"),
ylab = "Weight", xlab = "Groups")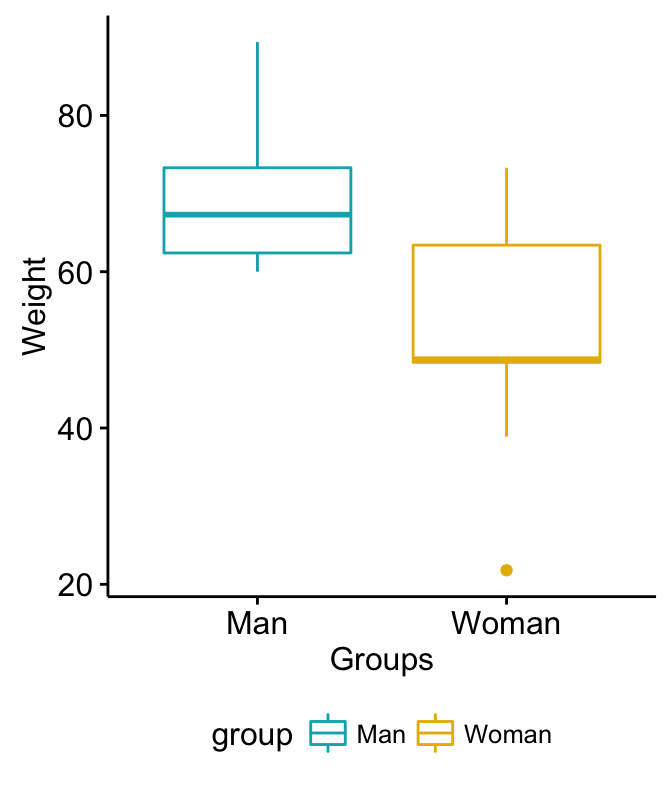
计算不成对的双样本秩检验
问题:女性和男性体重有显着差异吗?
1)计算双样本Wilcoxon检验 – 方法1:数据保存在两个不同的数值向量中。
res <- wilcox.test(women_weight, men_weight)
res
Wilcoxon rank sum test with continuity correction
data: women_weight and men_weight
W = 15, p-value = 0.02712
alternative hypothesis: true location shift is not equal to 0它将发出一条警告信息,称为“无法用平局计算精确的p值”。它可以通过添加另一个参数exact = FALSE来抑制此消息,但结果将是相同的。
2)计算双样本Wilcoxon检验 – 方法2:将数据保存在数据框中。
res <- wilcox.test(weight ~ group, data = my_data,
exact = FALSE)
res
Wilcoxon rank sum test with continuity correction
data: weight by group
W = 66, p-value = 0.02712
alternative hypothesis: true location shift is not equal to 0# 输出 p-value
res$p.value[1] 0.02711657如您所见,这两种方法给出了相同的结果。
测试的p值为 0.02712,小于显着性水平α= 0.05。我们可以得出结论,男性的中位数体重与女性的中位数体重显着不同,p值 = 0.02712。
注意:
- 如果你想测试男性体重的中位数是否小于女性体重的中位数,请输入:
wilcox.test(weight ~ group, data = my_data,
exact = FALSE, alternative = "less")- 或者,如果您想测试男性体重的中位数是否大于女性体重的中位数,请输入此值
wilcox.test(weight ~ group, data = my_data,
exact = FALSE, alternative = "greater")可下载资源
关于作者
Kaizong Ye是拓端研究室(TRL)的研究员。在此对他对本文所作的贡献表示诚挚感谢,他在上海财经大学完成了统计学专业的硕士学位,专注人工智能领域。擅长Python.Matlab仿真、视觉处理、神经网络、数据分析。
本文借鉴了作者最近为《R语言数据分析挖掘必知必会 》课堂做的准备。
非常感谢您阅读本文,如需帮助请联系我们!

 视频讲解|Stata和R语言自助法Bootstrap结合GARCH对sp500收益率数据分析
视频讲解|Stata和R语言自助法Bootstrap结合GARCH对sp500收益率数据分析 高维变量选择专题|R、Python用HOLP、Lasso、SCAD、PCR、ElasticNet实例合集分析企业财务、糖尿病、基因数据
高维变量选择专题|R、Python用HOLP、Lasso、SCAD、PCR、ElasticNet实例合集分析企业财务、糖尿病、基因数据 R语言汇率、股价指数与GARCH模型分析:格兰杰因果检验、脉冲响应与预测可视化
R语言汇率、股价指数与GARCH模型分析:格兰杰因果检验、脉冲响应与预测可视化 R语言分析糖尿病数据:多元线性模型、MANOVA、决策树、典型判别分析、HE图、Box’s M检验可视化
R语言分析糖尿病数据:多元线性模型、MANOVA、决策树、典型判别分析、HE图、Box’s M检验可视化


