在当今的医学领域,乳腺癌作为一种严重威胁女性健康的疾病,其治疗一直是科研工作者们关注的焦点。
乳腺癌的发展与雌激素受体密切相关,其中 ERα 被视为治疗乳腺癌的重要靶标。
能够拮抗 ERα 活性的化合物有望成为治疗乳腺癌的候选药物。然而,药物筛选是一个复杂而艰巨的过程。
一方面,在传统的药物筛选中,面临着诸多难点。各类化合物具有繁多的特征属性,如 ADMET 特性和化合物生物活性等。
如何从众多特征中挑选出影响化合物活性的关键特征,以及确定这些关键特征的取值范围以保证药物具有较高的生物活性,同时在 ADMET 五个特性中至少有三个表现良好的情况下使化合物活性最大化,这些都是极具挑战性的问题。
另一方面,随着科技的不断进步,人工智能和机器学习技术为药物研发带来了新的机遇。主成分分析、机器学习和多目标优化等方法在药物筛选中展现出了巨大的潜力。通过这些方法,可以有效地处理复杂的药物数据,挖掘出关键特征,预测药物的生物活性和参数范围,实现多目标的优化。
与此同时,量子计算的发展也为药物发现提供了新的思路。量子生成对抗网络(Quantum GAN)结合了量子计算和生成对抗网络的优势,能够处理高维数据,为药物研发提供了新的途径。通过量子电路和经典神经网络的结合,有望生成具有特定药物特性的分子,为新药研发带来新的突破。
本文将围绕主成分分析、机器学习和多目标优化在化合物特征药物筛选中的应用,以及量子生成对抗网络在药物发现(附数据代码)中的研究展开,旨在探索更加高效、准确的药物筛选和发现方法,为乳腺癌的治疗提供新的候选药物。
主成分分析、机器学习、多目标优化对化合物特征进行药物筛选
乳腺癌的发展与雌激素受体密切相关。目前抗激素治疗常用于ERα表达的乳腺癌患者。因此ERα被认为是治疗乳腺癌的重要靶标,能够拮抗ERα活性的化合物可能是治疗乳腺癌的候选药物。
在该项目中出现了各种难点,例如,各类化合物的特征属性繁多,比如有ADMET(Absorption,Distribution,Metabolism,Excretion,Toxicity)特性和化合物生物活性等。该如何选择关键特征是大问题。
选择了关键特征,如何确定该特征的取值大小才能保证该药品具有生物活性,首先要保证药物有效,才能讨论药性。
针对五个关键的特性ADMET如何在尽量让五个特性表现良好的情况下,让化合物活性尽量的高,这样的化合物的分子描述符的取值范围的寻找较为困难。
Chenhao Wu
可下载资源
9.Python用RNN循环神经网络:LSTM长期记忆、GRU门循环单元、回归和ARIMA对COVID-19新冠疫情新增人数时间序列预测
解决方案
任务/目标
- 在众多特征中找到影响化合物活性的关键特征。
- 找到关键特征的取值范围使得药物的生物活性较高。
- 找到在使得药效达标(ADMET五个特性至少有三个表现良好)的情况下生物活性较高
数据源准备
该项目的数据源由其他企业提供
数据清洗
视频
LSTM神经网络架构和原理及其在Python中的预测应用
视频
【视频讲解】神经网络、Lasso回归、线性回归、随机森林、ARIMA股票价格时间序列预测
视频
Python、R时间卷积神经网络TCN与CNN、RNN预测时间序列实例
视频
卷积神经网络CNN肿瘤图像识别
视频
CNN(卷积神经网络)模型以及R语言实现
视频
【视频讲解】Python深度学习股价预测、量化交易策略:LSTM、GRU深度门控循环神经网络附代码数据
视频
ResNet深度学习神经网络原理及其在图像分类中的应用Python代码
在数据中将大量字段为空的药物删除,少量字段为空的补0。针对第一个问题给他贴好标签,将化合物活性贴好标签,分为1-10,10个等级,在5以上的为活性较高,5以下的活性较低,更进一步,在5以上就贴上1,在5以下就贴上0。便于后续对其进行主成分分析。
针对第二个难题,是一个定量预测问题。我们将数据中需要的药物提取出来。
第三个问题是一个多目标优化的问题。我们同样给药物贴好标签,在ADMET属性中,满足3个或以上的我们给他贴1标签,否则贴0标签,后续对其进行多目标优化。
构造模型
针对问题1.我们使用了不同的主成分分析,包括相关性分析,随机森林 特征选择,基于相关性的特征子集选择算法,基于分类器的属性价值评估算法,Relief 算 法,基于分类器的特征子集选择算法,信息增益排序等。发现RReliefF算法更优秀,这里我们选择了RReliefF方法。
第二个问题采用了多种机器学习方法。包括极限随机树(ET),多层感知机(MLP),支持向量机回归(SVR),并对其进行模型评价,发现ET效果最好,并可以预测所有参数的范围。
第三个问题是多目标优化的问题,针对这类问题,我们使用的是粒子群算法,通过调节超参,达到近似全局最优解。
划分训练集和测试集
所有的药物是独立的样本,所以我们直接采用十折交叉验证即可。
上线之后的迭代,在得到所有化合物参数性能指标之后,我们要交给专业人士去临床验证这些药物。
部分模型预测结果:





该项目涉及到分类模型,回归模型,优化问题。是个综合的项目,都达到了不错的性能。
Python量子生成对抗网络QGAN神经网络的药物发现研究|附数据代码
开发新药是一项极具挑战性的工作,传统方法耗时漫长且成功率较低。随着人工智能和机器学习的发展,为新药研发带来了新的机遇。本文介绍了一种基于量子生成对抗网络(Quantum GAN,QGAN)的方法在药物发现中的应用,通过利用量子计算的优势处理高维数据,为药物研发提供了新的思路和方法。
一、引言
新药开发是一个复杂而漫长的过程,从靶点发现到临床试验,往往需要数年甚至数十年的时间。而且,经过重重筛选和测试,最终能够成功上市的药物寥寥无几。近年来,人工智能和机器学习技术在各个领域取得了显著的进展,也为药物研发带来了新的希望。其中,生成对抗网络(GAN)在图像生成等领域表现出色,而将量子计算与 GAN 相结合,有望在药物发现中发挥更大的作用。
二、量子相关术语
(一)量子电路
量子电路是由一系列门组成的有序集合,用于改变量子比特的状态以执行特定的量子运算。
(二)量子噪声
量子噪声是指任何可能导致量子计算机出现故障的因素。当量子比特暴露于噪声源时,量子计算机中的信息会受到干扰和降级。
(三)量子 GAN
在量子 GAN 中,GAN 模型的生成器和判别器在量子计算机或设备上进行训练,这些设备能够轻松处理高维数据(即量子数据)。
(四)带混合发生器的 Quantum GAN
QGAN-HG 由参数化量子电路组成,可提供量子比特大小维度的特征向量。需要一个经典的神经网络来生成所需的原子向量和键矩阵,以构建分子的图形表示,其中节点表示原子,边表示键。
三、QGAN量子电路
该电路在量子层的帮助下提供特征向量,其计算时间为 O(poly(log(M)))。量子层由初始化、参数化和测量三个阶段组成。在初始化部分,从 [-π, π] 中均匀采样两个参数 z1 和 z2,然后将其转换为数学形式,并对所有层重复操作,以生成酉矩阵 U(θ)。最后,通过对最终量子态应用量子运算来获取特征向量。图中提到的 Ry 和 Rz 是旋转门。
随时关注您喜欢的主题

四、QGAN-神经网络
从 QGAN电路生成特征向量后,这些向量被输入到经典神经网络中。此神经网络的输出包含原子层和键层,用于生成原子向量和键矩阵。
五、用于计算的指标
(一)Frechet 距离
它用于测量真实分子和合成分子分布之间的相似性。
(二)药物特性
药物性质包括药物可能性、溶解度、合成性等。这些特性与其他属性一起使用 RDKit 进行测量。
六、模型架构
如图所示:

- 第一张图片由药物片段和结合位点(受体)组成。药物的形状若适合受体,则有可能治愈疾病,这可以用锁和钥匙的概念来解释,受体如同锁,药物如同钥匙。
- 第二张图像由量子阶段和经典阶段组成,以虚线分隔。
- 第三张图片由原子和键矩阵组成,用于构建合成分子的图形结构。
- 在最后一步中,将真实分子和合成分子输入经典判别器以区分两者,并使用 RDKit 包评估 Frechet 距离和药物特性。最终预测将反馈到两个神经网络和量子电路,以便在每个训练周期更新所有参数。
七、用于训练模型的数据集
本研究使用的数据集由 134K 个稳定的有机小分子和多达 9 个重原子组成。
八、依赖
此模型依赖于以下框架:
- python>=3.5
- pytorch>=0.4.1
九、模型实现
(一)导入所需库和包
首先,导入所有必需的库和包,为后续的模型构建和训练做准备。
(二)设置量子比特单元并生成量子电路
初始化两个随机均匀噪声参数 z1 和 z2,然后为原子向量和节点矩阵生成一个电路。
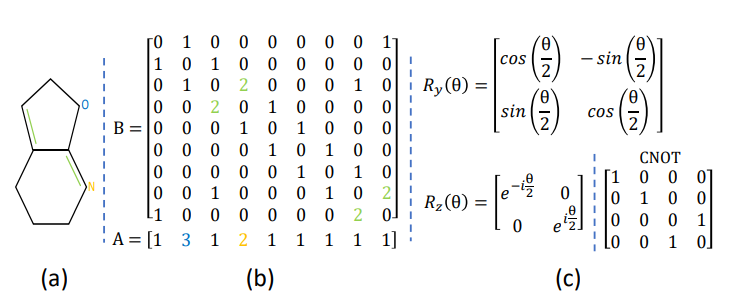
以下是生成量子电路的代码及解释
# 这里 a 是邻接矩阵,x 是节点
mols, _, _, a, x, _, _, _, _ = self.data.next_train_batch(self.batch_size)
a = torch.from_numpy(a).to(self.device).long() # 邻接矩阵。
x = torch.from_numpy(x).to(self.device).long() # 节点。
# "将标签索引转换为独热向量
a_tensor = self.label2onehot(a, self.b_dim)
x_tensor = self.label2onehot(x, self.m_dim)
z = torch.stack(tuple(ibm_sample_list)).to(self.device).float()
# Z-to-target
# 计算假图像的损失。
edges_logits, nodes_logits = self.G(z)
# 用 Gumbel softmax 进行后处理
(edges_hat, nodes_hat) = self.postprocess((edges_logits, nodes_logits), self.post_method)
logits_fake, features_fake = self.D(edges_hat, None, nodes_hat)
g_loss_fake = - torch.mean(logits_fake)
# 计算真图像的损失
# 真实奖励
rewardR = torch.from_numpy(self.reward(mols)).to(self.device)
# 假图像奖励
(edges_hard, nodes_hard) = self.postprocess((edges_logits, nodes_logits), 'hard_gumbel')
edges_hard, nodes_hard = torch.max(edges_hard, -1)[1], torch.max(nodes_hard, -1)[1]
mols = [self.data.matrices2mol(n_.data.cpu().numpy(), e_.data.cpu().numpy(), strict=True)
for e_, n_ in zip(edges_hard, nodes_hard)]
rewardF = torch.from_numpy(self.reward(mols)).to(self.device)
# 价值损失
value_logit_real,_ = self.V(a_tensor, None, x_tensor, torch.sigmoid)
value_logit_fake,_ = self.V(edges_hat, None, nodes_hat, torch.sigmoid)
g_loss_value = torch.mean((value_logit_real - rewardR) ** 2 + (
value_logit_fake - rewardF) ** 2)这段代码主要进行模型的推理和损失计算。首先从数据集中获取训练批次的数据,然后对生成器和判别器的输出进行处理,计算真假图像的损失和价值损失,并通过 Frechet 距离计算键和原子的距离损失,最后将这些损失存储在一个字典中。
(五)分子评估
最后一步是通过 RDKit 评估所有分子,并输出评估结果。

结论
本文介绍了一种基于量子生成对抗网络的药物发现方法,通过量子电路和经典神经网络的结合,能够生成具有特定药物特性的分子。该方法在数据集上进行了训练和测试,取得了较好的结果。然而,该方法仍处于研究阶段,需要进一步的改进和优化。未来,可以通过增加数据集的规模、改进模型架构和优化算法等方式,提高模型的性能和泛化能力,为药物发现提供更有力的支持。

关于分析师
Chenhao Wu
在此对 Chenhao Wu 对本文所作的贡献表示诚挚感谢,他在上海海事大学完成了计算机应用技术专业的硕士学位,专注机器学习、数据处理领域。擅长 WEKA、Python、Java。

 SPSS与Python用Resblock优化BP神经网络分析慢性胃炎病历数据聚类K-means/AGNES、关联规则挖掘及预测
SPSS与Python用Resblock优化BP神经网络分析慢性胃炎病历数据聚类K-means/AGNES、关联规则挖掘及预测 Python神经网络、随机森林、PCA、SVM、KNN及回归实现ERα拮抗剂、ADMET数据预测|附代码数据
Python神经网络、随机森林、PCA、SVM、KNN及回归实现ERα拮抗剂、ADMET数据预测|附代码数据 Python农作物种植策略研究GA-BP神经网络、蒙特卡洛算法、自注意力Stacking集成模型及粒子群算法PSO优化基于华北山区乡村农作物数据及地块数据
Python农作物种植策略研究GA-BP神经网络、蒙特卡洛算法、自注意力Stacking集成模型及粒子群算法PSO优化基于华北山区乡村农作物数据及地块数据 MATLAB奥运会奖牌预测研究 —CNN神经网络、逻辑回归、Liang-Kleeman信息流、多元回归及随机森林模型的因果关联与概率预测|附代码数据
MATLAB奥运会奖牌预测研究 —CNN神经网络、逻辑回归、Liang-Kleeman信息流、多元回归及随机森林模型的因果关联与概率预测|附代码数据



