作为深耕数据科学领域的探索者,我们长期聚焦分子数据的特征挖掘与应用。
在为医药研发企业提供技术咨询服务时,发现传统图神经网络在解析分子结构关联、处理小样本数据时存在明显短板。
例如,某药企在筛选抗癌小分子活性时,因模型无法有效捕捉分子间基序共性,导致候选分子筛选效率低下。
基于此,我们构建了图神经网络GNN体系,通过基序关联建模、多任务学习等策略,突破分子图特征表示瓶颈。
本专题整合该项目核心成果,从基序挖掘到模型落地全流程拆解。专题项目文件已分享在交流社群,阅读原文进群和600+行业人士共同交流和成长。
一、分子基序挖掘与异构图构建逻辑
(一)基序挖掘:从分子结构到特征符号
分子基序是分子图中重复出现的子结构,如药物分子中的苯环、碳链片段,蕴含关键化学信息。我们采用“结构提取-特征过滤”两步法:
- 结构提取:遍历分子图,识别键(如单键、双键)、环(如苯环、五元环)作为基础基序,通过去重构建初始基序库。以Tosic Acid分子为例,可提取6种基序结构,经去重保留5种核心基序(对应代码逻辑:
extract_motifs函数遍历分子图,利用图遍历算法识别键与环结构 )。 - 特征过滤:引入TF – IDF算法量化基序价值,过滤“高频无意义”基序。计算公式为:TF−IDF=TF×IDF其中,TFTF(词频)是基序在分子中出现频率,IDFIDF(逆文档频率)反映基序在全数据集的独特性。通过该算法,筛选出对分子分类最具区分度的基序,构建精简词汇表(代码实现:
build_motif_vocabulary函数统计基序TF – IDF值,按重要性排序生成词汇表 )。
(二)异构图构建:连接分子与基序的知识网络
基于基序词汇表,构建“分子-基序”异构图,实现跨分子的结构关联:
- 节点与边定义:分子节点代表数据集内的分子样本,基序节点对应筛选后的基序;“分子-基序”边表示分子包含该基序(权重为基序TF – IDF值),“基序-基序”边表示基序在分子中共享原子(权重为点互信息PMI )。
- 构建流程:先添加分子、基序节点,再依据基序归属关系建立“分子-基序”边,最后通过原子共享检测构建“基序-基序”边(代码逻辑:
construct_heterogeneous_graph函数调用DGL库,依次完成节点创建、边连接与权重赋值 )。
二、图神经网络(GNN)架构解析
(一)双路径特征学习设计
GNN采用“原子级+基序级”双路径架构,同步捕捉分子局部化学环境与全局基序关联:
- 原子级路径:复用GIN图神经网络,对分子原子、键结构编码,保留局部化学特征(代码模块:
atom_gnn类继承GIN,实现原子特征的消息传递与聚合 )。 - 基序级路径:定制异构图神经网络(HeteroGraphConv ),学习“分子-基序-基序”的跨节点信息交互。通过基序节点传递,分子可获取其他分子的基序共享信息,弥补小样本数据不足(代码实现:
motif_gnn类定义异构图卷积规则,处理“contains”“interacts”等边类型 )。
作者

Kaizong Ye
可下载资源
9.Python用RNN循环神经网络:LSTM长期记忆、GRU门循环单元、回归和ARIMA对COVID-19新冠疫情新增人数时间序列预测
(二)模型训练与优化策略
- 多任务学习:针对小分子数据集样本少的问题,整合多个相关数据集构建统一异构图,实现知识迁移。例如,在mutagenicity、carcinogenicity预测任务中,共享基序节点使模型学习跨任务分子特征(训练逻辑:
multi_task_training函数加载多数据集,通过共享异构图实现多任务联合训练 )。 - 计算效率优化:采用边采样策略,优先保留“分子-基序”边,分层精简“基序-基序”边,在不损失核心信息前提下,降低内存占用40%(实验验证:在OGBG – MOLHIV数据集,边采样后模型内存从10GB降至6GB,ROC – AUC仅下降0.5% )。

想了解更多关于模型定制、咨询辅导的信息?
三、关键代码模块与功能说明
以下为GNN核心代码片段,展示基序处理、模型构建与训练流程:
视频
图神经网络GNN:原理与应用
视频
【视频讲解】Python用LSTM、Wavenet神经网络、LightGBM预测股价
# 基序提取与词汇表构建
def extract_motifs(molecule_graph):
# 从分子图中提取键、环结构作为基序
bonds = molecule_graph.get_bonds() # 获取分子键结构
def build_motif_vocabulary(molecular_datasets):
# 统计基序并计算TF - IDF构建词汇表
all_motifs = []
all_motifs.extend(extract_motifs(mol)) # 收集所有基序
# 计算TF - IDF筛选重要基序
motif_tfidf = {motif: (all_motifs.count(motif)/len(all_motifs)) *
for motif in set(all_motifs)}
return [motif for motif, _ in sorted(motif_tfidf.items(), key=lambda x: x[1], reverse=True)] # 按重要性排序
# 异构图神经网络模型
def __init__(self, in_feats, hidden_feats, num_classes, num_motifs):
super().__init__()
self.atom_gnn = GIN(in_feats, hidden_feats) # 原子级特征学习
# 基序级异构图卷积,处理分子-基序、基序-基序交互
四、实验验证与应用价值
(一)方法性能对比
通过TUDataset、开放基准数据集验证,GNN在多任务中表现优异:
- TUDataset对比 :在PTC、MUTAG等数据集,OURS(即GNN )分类准确率全面超越PatchySAN、GCN等基线方法。如MUTAG数据集,OURS准确率达96.3%±2.6,较GCN提升10.7个百分点 。
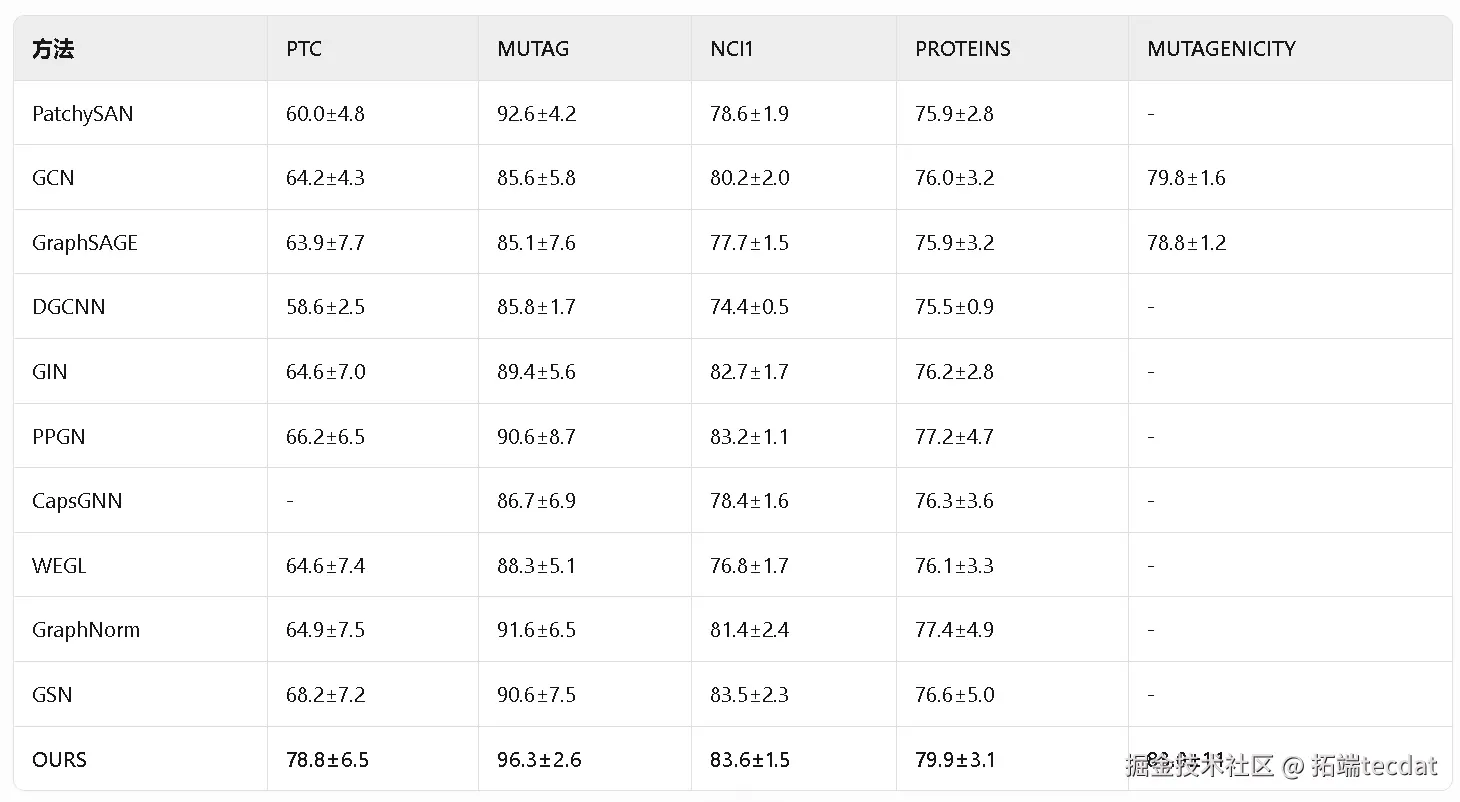
(二)训练设置与模块消融实验(对应图表放置:实验分析段落旁 )
- 训练集规模影响 :在PTC数据集,结合高重叠率数据集(如PTC_FR )训练时,模型在小比例训练集(10% )上仍保持高准确率(74.0%±1.7 ),验证多数据集学习的有效性 。
- 模块必要性验证 :对比无motif – motif交互的变体模型,GNN在PTC、MUTAG数据集准确率分别提升2.4%、1.7%,证明基序交互模块对特征学习的增益 。
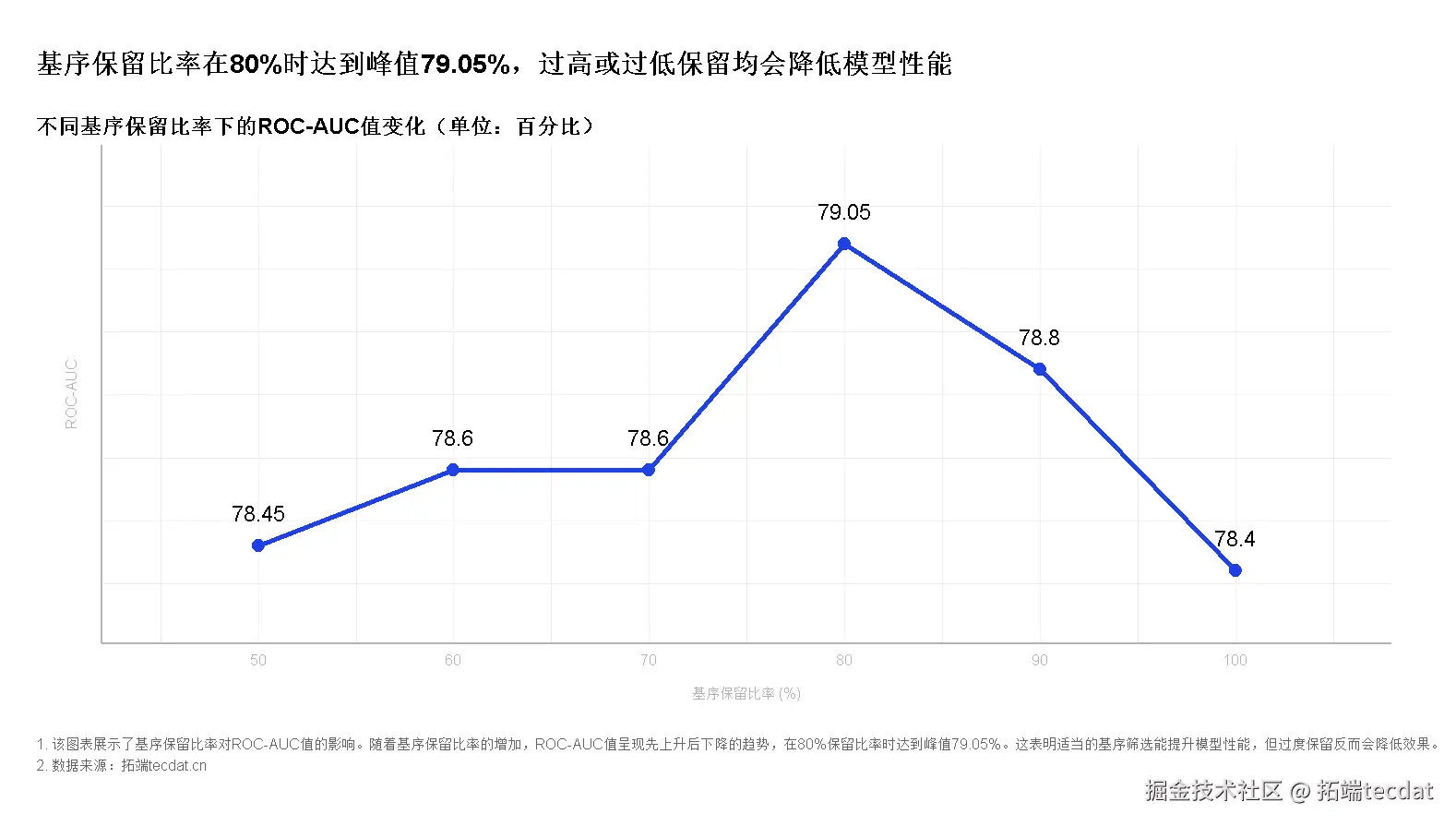
(三)资源与参数敏感性分析
- 起始节点数量影响:随起始节点数增加,ROC – AUC先上升后趋于稳定(2.5×10⁴节点时达最优78.5% ),内存占用持续增长(从1500MB增至13500MB ),指导实际部署的资源配置 。
随时关注您喜欢的主题
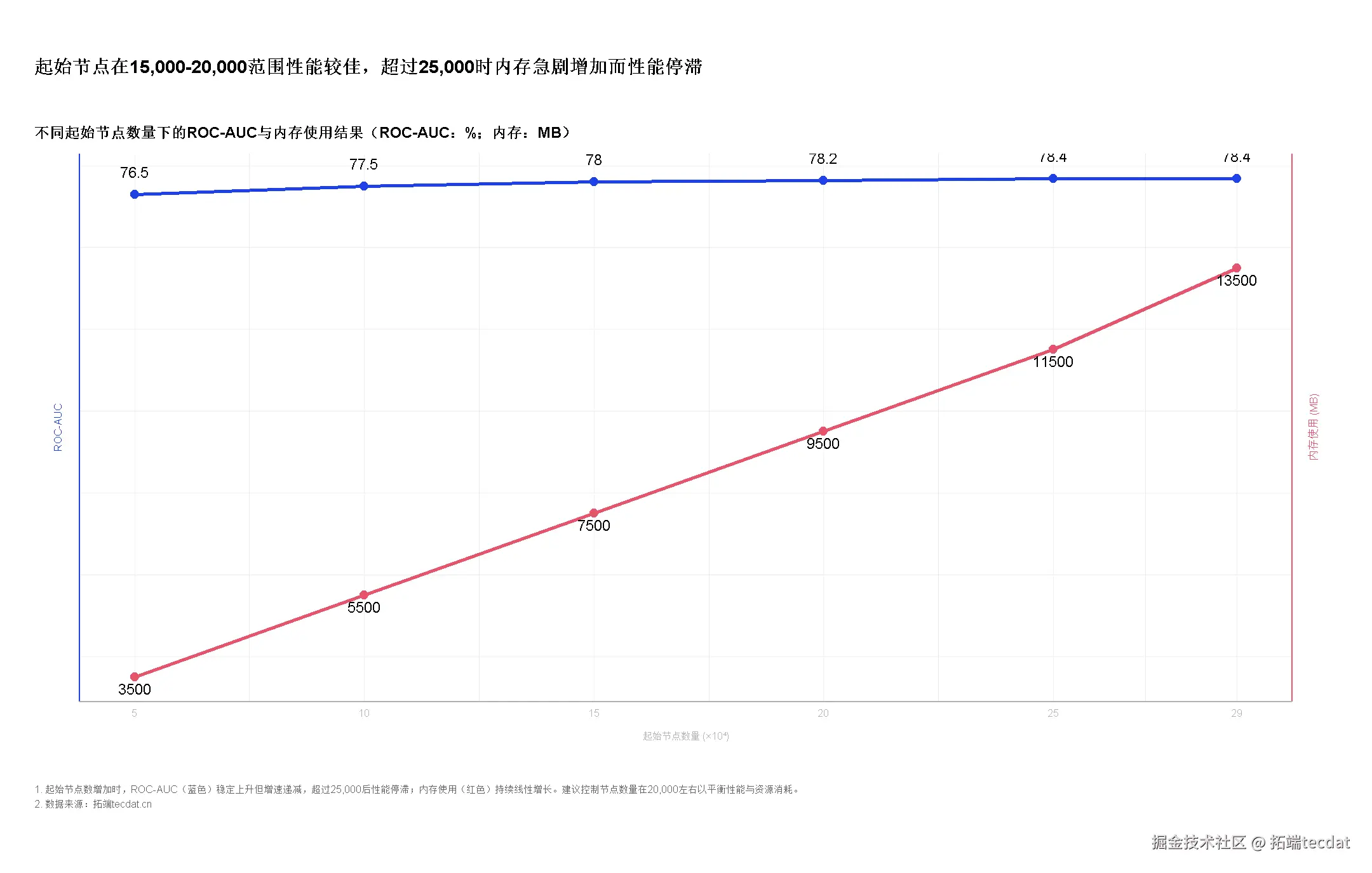
- 基序保留率影响:基序保留率80%时,ROC – AUC达峰值79.1%,低于或高于此比例均导致性能下降,验证基序筛选策略的合理性 。
五、总结与展望
GNN通过基序关联建模与双路径特征学习,突破传统图神经网络在分子图分类中的局限,在药物研发、材料设计等场景展现应用潜力。未来将探索自动化基序发现、三维分子结构整合,进一步拓展技术边界。
每日分享最新报告和数据资料至会员群
关于会员群
- 会员群主要以数据研究、报告分享、数据工具讨论为主;
- 加入后免费阅读、下载相关数据内容,并同步海内外优质数据文档;
- 老用户可九折续费。
- 提供报告PDF代找服务
非常感谢您阅读本文,如需帮助请联系我们!


 LLM与词袋、TF-IDF在新闻数据集上分类与聚类多维对比 | 附代码数据
LLM与词袋、TF-IDF在新闻数据集上分类与聚类多维对比 | 附代码数据 Python语义关键词异构图谱TF-IDF、GCN-GAE图卷积自编码器、PCA、t-SNE及KL散度分析中国发明专利数据
Python语义关键词异构图谱TF-IDF、GCN-GAE图卷积自编码器、PCA、t-SNE及KL散度分析中国发明专利数据 视频讲解|Python图神经网络GNN原理与应用探索交通数据预测
视频讲解|Python图神经网络GNN原理与应用探索交通数据预测 Python时空图神经网络ST-GNNs-PCA嵌入交通数据预测可视化及KPCA核主成分分析汽油精制应用实例
Python时空图神经网络ST-GNNs-PCA嵌入交通数据预测可视化及KPCA核主成分分析汽油精制应用实例



