大语言模型高级Prompt临床科研辅助研究——AdaBoost、LightGBM、MLP等模型的食道癌预测、遗传性听力损失诊断及心肌病识别
从数据科学视角来看,临床科研的核心价值在于通过数据挖掘与分析转化为可落地的诊疗优化方案,但当前临床科研领域普遍面临”技术门槛高、效率低”的行业痛点。
本项目报告、代码和数据资料已分享至会员群
专题名称:大语言模型Prompt工程驱动的临床科研数据智能分析专题
临床工作者往往具备深厚的医学专业积累,却缺乏系统的数据科学训练,难以熟练运用机器学习工具完成科研数据分析全流程;而传统数据分析工具如Python、R等,对非专业用户的学习成本过高,严重制约了临床科研的推进效率。
随着大语言模型技术的快速发展,其在自然语言理解、代码生成等领域的能力为临床科研数字化转型提供了新路径。但通用大语言模型在医学场景应用中存在明显短板,如提示词设计专业性不足、输出结果缺乏医学适配性等,这成为制约技术落地的关键瓶颈。
本文内容改编自过往客户咨询项目的技术沉淀并且已通过实际业务校验,该项目完整代码与数据已分享至交流社群。阅读原文进群,可与800+行业人士交流成长;还提供人工答疑,拆解核心原理、代码逻辑与业务适配思路,帮大家既懂 怎么做,也懂 为什么这么做;遇代码运行问题,更能享24小时调试支持。
本专题围绕”大语言模型Prompt工程适配临床科研”核心需求,构建高级Prompt生成器,结合逻辑回归(LR)、自适应提升(AdaBoost)、轻量梯度提升机(LightGBM)、极端梯度提升(XGBoost)、随机森林(RF)、支持向量机(SVM)、决策树(DT)、k-最近邻(KNN)、多层感知器(MLP)等多种数据分析模型,针对食道癌预测、遗传性听力损失诊断、野生型甲状腺素蛋白淀粉样变性心肌病识别三类典型临床科研场景开展实践验证,系统阐述技术方案的设计思路、实现流程与应用效果,为临床工作者提供低门槛、高适配的科研数据分析解决方案。
本项目报告、代码和数据资料
文章脉络流程图(竖版)
项目文件目录截图
课题研究背景和意义
医学研究是推动临床实践进步的核心动力,也是提升患者诊疗水平的关键环节。但在当前临床科研实践中,临床工作者面临诸多现实挑战:临床科研涵盖研究设计、数据采集、统计分析、模型构建、结果解释等多个环节,既要求具备医学专业知识,还需掌握统计学基础和编程能力。而临床工作者大多缺乏系统的数据科学训练,尤其在机器学习建模、大数据分析等领域存在明显知识断层。
现有数据分析工具如Python、R、SPSS等,均要求用户具备一定的编程或软件操作能力,非专业用户即便完成简单的数据清洗或回归分析,也需投入大量时间学习技术细节,导致科研效率低下。尽管已有部分医学研究辅助软件,如文献管理工具、统计向导工具等,但这些工具功能局限于单一任务,无法为复杂科研流程提供端到端支持。如何将人工智能技术深度融入科研全流程,成为行业亟待解决的难题。
通用型人工智能工具虽具备一定自然语言处理能力,但生成的代码或分析建议往往缺乏医学领域针对性,存在术语不准确、方法不专业等问题,难以直接满足临床科研需求。在此背景下,开发一款能够降低技术门槛、适配医学领域需求、覆盖科研全流程的智能辅助系统,已成为提升临床科研效率的迫切需求。
近年来,以GPT-4、DeepSeek为代表的大语言模型(LLMs)通过海量文本预训练和指令微调,展现出强大的自然语言理解、代码生成和逻辑推理能力,为医学智能化提供了新可能。在医学领域,大语言模型的应用潜力主要体现在三个方面:一是文献解析与知识关联,可快速解析医学文献、临床指南和病例报告,生成文献综述框架或提取关键研究结论;二是研究设计辅助,能基于研究目标推荐合适的研究设计类型和统计分析方法;三是代码生成与结果解读,通过自然语言指令自动生成数据预处理、可视化分析或机器学习建模的代码脚本,还可针对统计分析结果生成符合学术规范的解读文本。
但现有大语言模型在医学场景应用中仍存在显著局限性:提示词依赖性强,输出质量高度依赖输入提示的设计,非专业用户难以构造精准的医学任务指令;领域适配不足,通用模型缺乏对医学专业术语、研究范式的深度理解,易产生逻辑错误或方法误导;交互效率低下,缺乏针对复杂任务的渐进式交互机制,用户需反复调试提示词才能获得理想输出。这些问题的核心在于如何通过提示词工程实现大语言模型与医学场景的高效适配,这也是本研究的核心重点。
本研究聚焦”基于大语言模型的临床科研辅助方法”开发,以提示词设计工程为核心突破点,具备重要的理论和实践价值。通过构建智能化科研辅助系统,将大语言模型深度嵌入研究设计、数据分析、结果解释等全流程,实现”自然语言驱动科研”。临床工作者可直接简单描述需求,系统自动生成统计代码、可视化图表与方法学建议,显著降低技术工具使用门槛,推动医学研究范式革新,解决提示词设计的医学适配难题。
最受欢迎的见解
- Python员工数据人力流失预测:ADASYN采样CatBoost算法、LASSO特征选择与动态不平衡处理及多模型对比研究
- R分布式滞后非线性模型DLNM分析某城市空气污染与健康数据:多维度可视化优化滞后效应解读
- Python古代文物成分分析与鉴别研究:灰色关联度、岭回归、K-means聚类、决策树分析
- Python TensorFlow OpenCV的卷积神经网络CNN人脸识别系统构建与应用实践
- Python用Transformer、SARIMAX、RNN、LSTM、Prophet时间序列预测对比分析用电量、零售销售、公共安全、交通事故数据
- MATLAB贝叶斯超参数优化LSTM预测设备寿命应用——以航空发动机退化数据为例
- Python谷歌商店Google Play APP评分预测:LASSO、多元线性回归、岭回归模型对比研究
- Python+AI提示词糖尿病预测模型融合构建:伯努利朴素贝叶斯、逻辑回归、决策树、随机森林、支持向量机SVM应用
软件的测试、验证和评价
测试、验证和评价方案
本研究通过对比大语言模型生成的研究结果与临床已发表论文报道的研究结果,验证系统的实际应用有效性。选取食道癌预测、变异遗传性听力损失诊断、野生型甲状腺素蛋白淀粉样变性心肌病识别三类典型临床科研场景开展验证,采用AUROC值、平均精度、F1分数等核心指标进行性能评估。
食道癌的预测

本研究通过多维度验证体系确保技术方案的可靠性,除核心性能指标外,还结合临床专家评审、实际应用反馈等维度综合评估系统效果,确保输出结果符合临床科研的专业要求和实际应用场景的需求。
任务说明
食管鳞状细胞癌和食管胃交界处腺癌预后较差,早期发现是降低死亡率的关键。但早期发现依赖上消化道内窥镜检查,难以在人群水平上实施。本场景旨在构建基于机器学习的全自动预测工具,集成微创海绵细胞学测试和流行病学危险因素,用于内窥镜检查前的癌症筛查。
测试与验证
基于已发表研究的食道癌预测任务,提取研究背景、任务说明、数据集信息、模型训练要求和目标输出等核心模块信息,将其输入"高级Prompt生成器"得到针对性提示词,再将该提示词输入DeepSeek生成对应的Python分析代码。模型性能主要通过受试者工作特征曲线下面积(AUROC)和平均精度衡量。
食道癌预测提示词的生成过程如下:

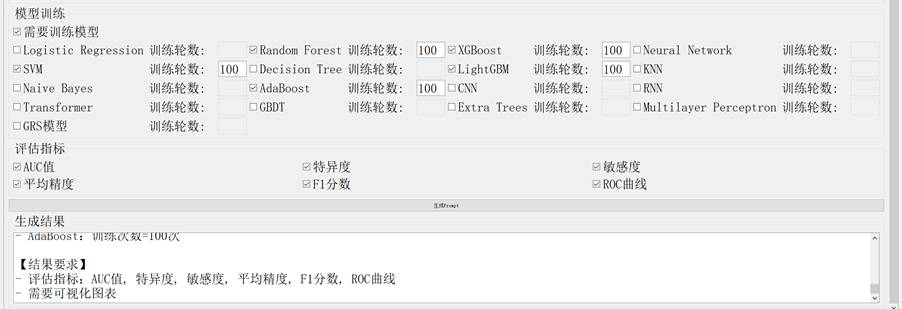
图2-1 食道癌预测Prompt生成图
对生成的Python代码进行文件路径修改,确保数据集正确读取。DeepSeek生成的模型在训练集上的性能表现如下表所示:
表2-1 DeepSeek生成的机器模型性能表现
| 机器学习模型 | AUC值 | 平均精度 |
|---|---|---|
| LightGBM | 0.9479 | 0.4286 |
| AdaBoost | 0.9523 | 0.3825 |
| XGBoost | 0.9403 | 0.4371 |
| SVM | 0.9329 | 0.4128 |
| Random Forest | 0.9448 | 0.3280 |
各模型在测试集中的AUROC值及其特征曲线如下:

图2-2 各模型在测试集中的AUROC值及其特征曲线
已发表研究中作者提供的模型测试结果如下:
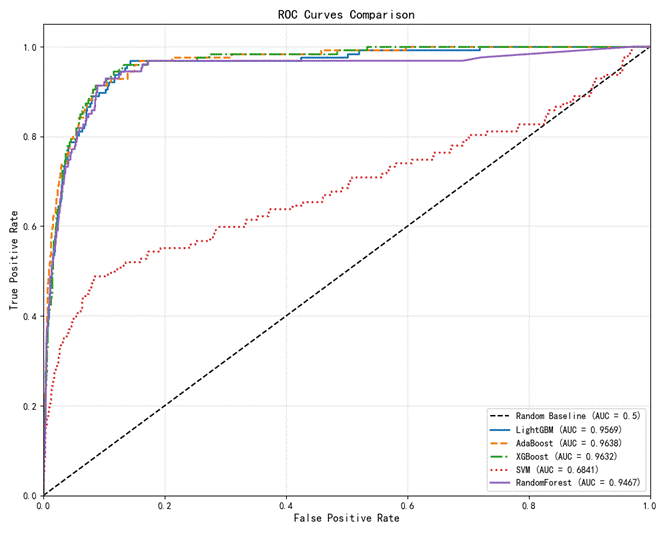
图2-3 原研究各模型的AUROC值及其特征曲线
核心代码实现(修改后)
# 数据准备阶段
# 读取训练集和测试集数据
train_data1 = pd.read_csv('./train.csv') # 原始训练集
train_data2 = pd.read_csv('./train_somte.csv') # SMOTE过采样后的训练集
train_data = pd.concat([train_data1, train_data2], axis=0) # 合并训练集
test_data = pd.read_csv('./test.csv') # 测试集
# 定义特征和目标变量
num_features = [f'f{i}' for i in range(1, 106)] # 数值特征(f1到f105)
cat_features = ['Sex', 'Age', 'Urban_rural', 'Smoker', 'SmokingIndex',
'SmokingExtent', 'Drinking', 'DrinkingExtent', 'Flush',
'Pickled', 'HotFood', 'TooothLoss', 'ToothLossNumber',
'GICancer', 'FamilyHistory'] # 分类特征
target_var = 'GroundTruth_bi' # 二分类目标变量
# 准备训练和测试数据集
X_train = train_data[num_features + cat_features]
y_train = train_data[target_var]
X_test = test_data[num_features + cat_features]
y_test = test_data[target_var]
# 数据预处理阶段
# 数值特征处理管道:均值填充缺失值 + 标准化
num_transformer = Pipeline(steps=[
('imputer', SimpleImputer(strategy='mean')),
('scaler', StandardScaler())
])
# 分类特征处理管道:众数填充缺失值 + 独热编码
cat_transformer = Pipeline(steps=[
('imputer', SimpleImputer(strategy='most_frequent')),
('onehot', OneHotEncoder(handle_unknown='ignore'))
])
# 组合处理器
preprocessor = ColumnTransformer(
transformers=[
('num', num_transformer, num_features),
('cat', cat_transformer, cat_features)
])
......注:上述代码省略了10次迭代训练中的预测及指标计算代码、ROC曲线绘制的核心代码,完整代码可通过交流社群获取。
结果对比
已发表研究中表现最佳的模型为LightGBM,其在测试集中的AUROC值为0.960。而DeepSeek生成的模型中,AdaBoost表现最优,AUROC值为0.952,与原始模型性能接近。
对比分析显示,DeepSeek生成的机器学习模型与重新实现的原始研究模型,AUROC值大致相似(分别为0.952和0.957),无显著差异,验证了生成代码的有效性。
变异遗传性听力损失的诊断
任务说明
遗传性听力损失是常见的感觉缺陷,严重影响患者生活质量。随着基因测序技术的发展,越来越多的变异被发现,但这些变异的遗传诊断对专业能力要求极高,人工诊断难度大。本场景旨在构建基于机器学习的遗传诊断模型,针对GJB2、SLC26A4和MT-RNR1三类遗传性听力损失相关变异进行诊断。
测试与验证
基于已发表研究的遗传性听力损失诊断任务,提取核心信息输入"高级Prompt生成器"得到提示词,再通过DeepSeek生成Python分析代码。模型性能通过AUROC值、准确性和F1分数进行评估。
遗传性听力损失诊断提示词的生成过程如下:

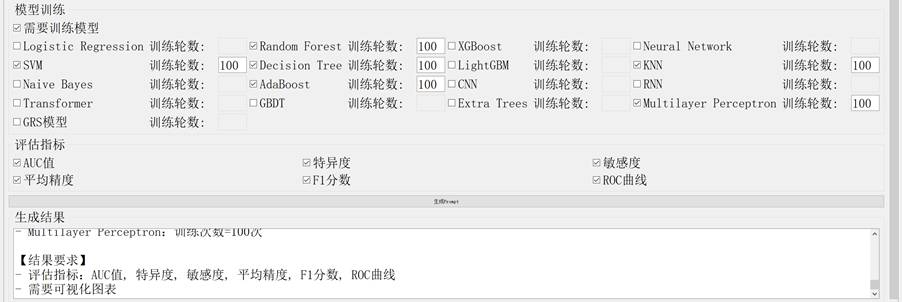
图2-4 HHL诊断Prompt生成图
对生成的Python代码进行文件路径修改后运行,DeepSeek生成的模型性能表现如下表所示:
表2-2 DeepSeek生成的机器模型性能表现
| 机器学习模型 | 准确率 | AUC值 | 平均精度 | F1分数 |
|---|---|---|---|---|
| Decision Tree | 0.728 | 0.660 | 0.848 | 0.837 |
| SVM | 0.764 | 0.700 | 0.855 | 0.866 |
| Random Forest | 0.766 | 0.682 | 0.885 | 0.868 |
| KNN | 0.773 | 0.718 | 0.862 | 0.850 |
| AdaBoost | 0.766 | 0.560 | 0.801 | 0.868 |
| MLP | 0.775 | 0.744 | 0.888 | 0.853 |
各模型在测试集中的AUROC值及其特征曲线如下:
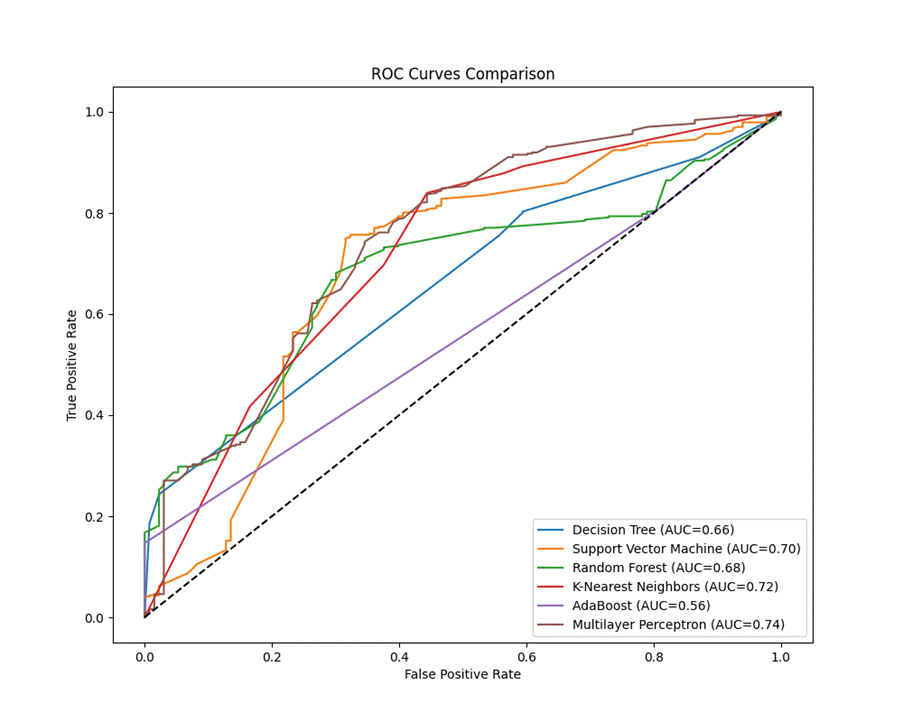
图2-5 各模型在测试集中的AUROC值及其特征曲线
核心代码实现(修改后)
def preprocess_data(train_path, test_path):
# 读取数据
train_df = pd.read_excel(train_path)
test_df = pd.read_excel(test_path)
# 定义基因相关特征列(省略具体列定义的详细代码)
gjb2_cols = ['p.F115C', 'p.M195V', ...] # 省略其他列
mtdna_cols = ['C1494T', 'A1555G', ...] # 省略其他列
slc26a4_cols = ['p.V22E', 'p.V207L', ...] # 省略其他列
all_features = gjb2_cols + mtdna_cols + slc26a4_cols
# 列对齐和数据清洗(省略详细代码)
def align_columns(df):
...
train_df = align_columns(train_df)
test_df = align_columns(test_df)
# 缺失值处理
imputer = SimpleImputer(strategy='most_frequent')
X_train = imputer.fit_transform(train_df[final_features])
y_train = train_df['Diagnoses'].values
X_test = imputer.transform(test_df[final_features])
y_test = test_df['Diagnoses'].values
return X_train, y_train, X_test, y_test
# 模型定义函数
def get_models():
return {
'Decision Tree': Pipeline([
('scaler', StandardScaler()),
('clf', DecisionTreeClassifier(max_depth=5, random_state=42))
]),
'Support Vector Machine': Pipeline([
('scaler', StandardScaler()),
('clf', SVC(kernel='rbf', C=1.0, probability=True, random_state=42))
]),
# 省略其他模型定义代码
'Multilayer Perceptron': Pipeline([
('scaler', StandardScaler()),
('clf', MLPClassifier(hidden_layer_sizes=(50,), max_iter=1000, random_state=42))
])
}
# 评估指标计算函数(省略详细代码)
def evaluate_model(model, X_test, y_test):
...
# 主程序
def main():
# 数据预处理
X_train, y_train, X_test, y_test = preprocess_data('./Discovery Set.xlsx', './Validation Set.xlsx')
# 获取模型集合
models = get_models()
# 初始化结果存储和绘图
cv_metrics = {}
test_metrics = {}
plt.figure(figsize=(10, 8))
# 模型训练与评估(省略循环训练的核心代码)
for name, model in models.items():
print(f"\n{'=' * 30}\nProcessing model: {name}\n{'=' * 30}")
# 交叉验证和模型训练
...
# 结果可视化
...
plt.show()
# 打印结果(省略详细代码)
...
if __name__ == "__main__":
main()
注:上述代码省略了特征列定义的详细代码、列对齐和数据清洗的详细代码、评估指标计算函数的详细代码、模型循环训练的核心代码,完整代码可通过交流社群获取。
结果对比
已发表研究中表现最佳的模型为SVM,AUROC值为0.751,且优于三位临床专家。DeepSeek生成的模型中,多层感知器(MLP)表现最佳,AUROC值为0.744,接近原始模型;但在准确率(0.775 vs 0.812)和F1分数(0.853 vs 0.861)方面略低于原始模型。
头对头分析显示,DeepSeek生成的最优模型AUROC值(0.744)优于重新实现的原始研究模型(0.616),存在较大差异。对比原始研究的模型性能曲线可见,DeepSeek生成的机器学习模型性能大部分优于原始研究方法,仅AdaBoost模型性能较差,可通过优化网格搜索参数进一步提升性能。
原始研究作者提供的模型测试结果如下:
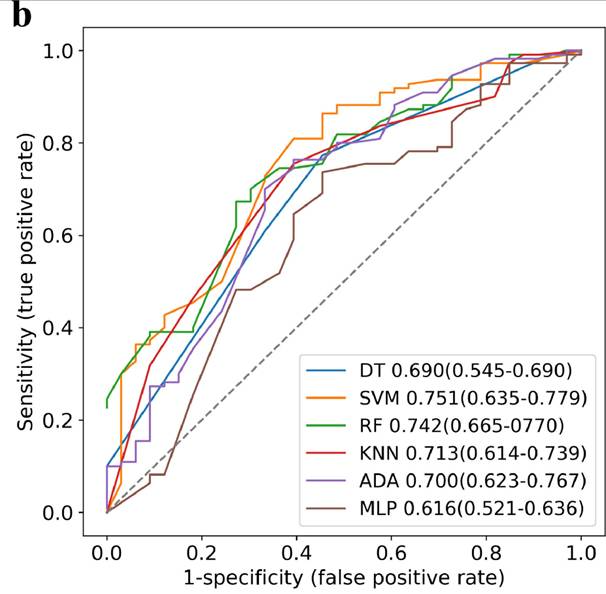
图2-6 原研究各模型的AUROC值及其特征曲线
野生型甲状腺素蛋白淀粉样变性心肌病的识别
任务说明
转甲状腺素蛋白淀粉样变性心肌病是心力衰竭的常见未识别原因,目前已有针对性治疗药物。因此,在不可逆性心力衰竭发生前,识别高危患者进行早期诊断和治疗至关重要。本场景旨在基于健康记录中的队列数据和已建立的医学诊断,构建机器学习模型识别有心脏淀粉样变性风险的患者。
测试与验证
基于已发表研究的心脏淀粉样变性识别任务,提取核心信息输入"高级Prompt生成器"得到提示词,通过DeepSeek生成Python分析代码。模型性能通过AUROC值、准确率和特异度进行评估。
心脏淀粉样变性识别提示词的生成过程如下:
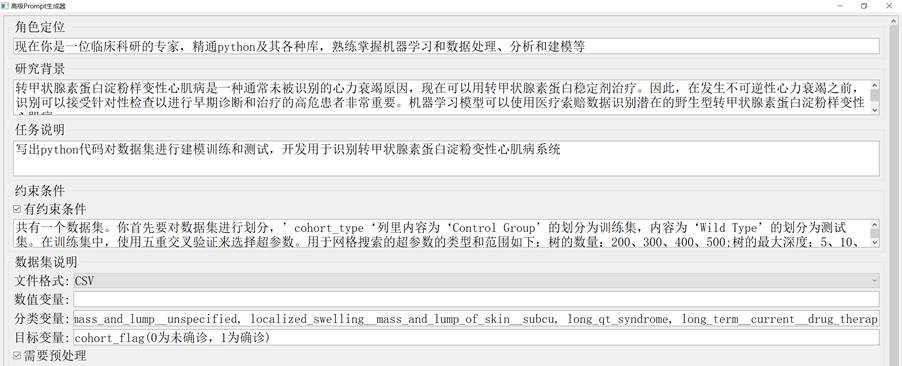
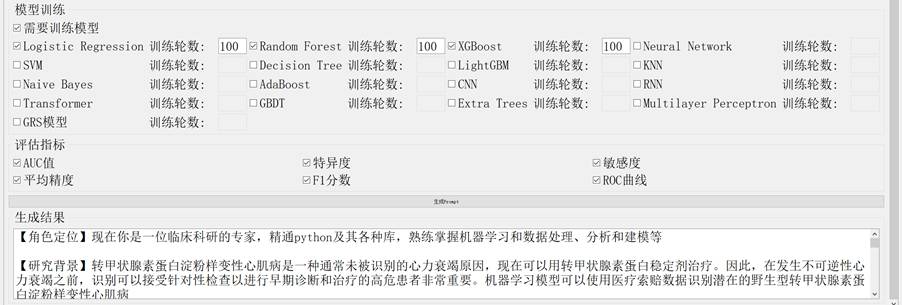
图2-7 心脏淀粉样变性识别Prompt生成图
对生成的Python代码进行文件路径修改后运行,结果如下表所示:
表2-3 DeepSeek生成的机器模型性能表现
| 机器学习模型 | AUC值 | 准确度 | 特异度 |
|---|---|---|---|
| Logistic Regression | 0.917 | 0.838 | 0.848 |
| XGBoost | 0.948 | 0.886 | 0.925 |
| Random Forest | 0.951 | 0.893 | 0.910 |
各模型在测试集中的AUROC值及其特征曲线如下:
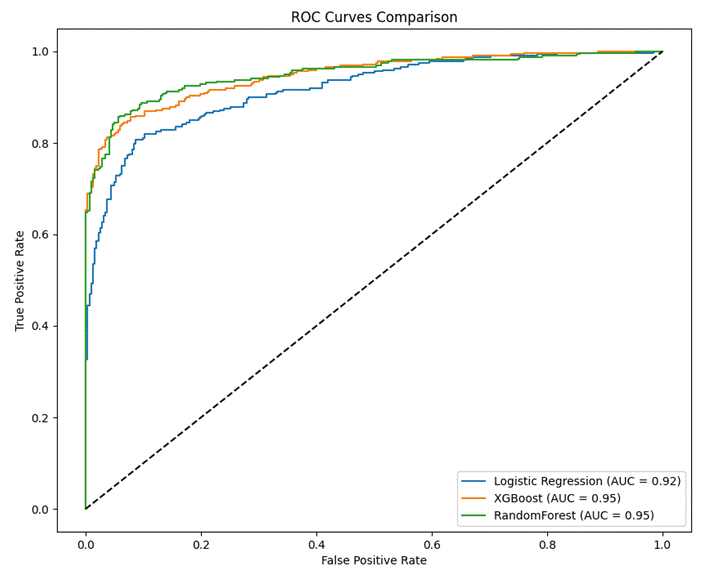
图2-8 各模型在测试集中的AUROC值及其特征曲线
核心代码实现(修改后)
# 导
# 数据读取
print("正在读取数据集...")
df = pd.read_csv('./data.csv')
# 数据划分与验证
print("\n=== 数据划分与验证 ===")
try:
# 按队列类型划分
train_df = df[df['cohort_type'] == 'Control Group']
test_df = df[df['cohort_type'] == 'Wild Type']
X_train = train_df.drop(columns=['cohort_flag', 'cohort_type'])
y_train = train_df['cohort_flag']
X_test = test_df.drop(columns=['cohort_flag', 'cohort_type'])
y_test = test_df['cohort_flag']
if len(np.unique(y_train)) < 2:
raise ValueError("训练集需要包含正负样本")
except Exception as e:
print(f"\n警告:{str(e)}")
print("正在启用备用划分方案:分层随机划分")
# 备用划分方案(省略详细代码)
X = df.drop(columns=['cohort_flag', 'cohort_type'])
y = df['cohort_flag']
X_train, X_test, y_train, y_test = train_test_split(
X, y, test_size=0.3, stratify=y, random_state=42
)注:上述代码省略了备用数据划分方案的详细代码、模型超参数的完整定义、超参数搜索与模型训练的核心代码、模型预测与评估的核心代码、结果可视化和表格展示的详细代码,完整代码可通过交流社群获取。
结果对比
已发表研究中,随机森林(RF)模型表现最佳,内部验证集AUROC值为0.930。DeepSeek生成的模型中同样是RF模型表现最优,且在AUROC值(0.951 vs 0.930)和准确率(89% vs 87%)指标上优于原始模型。
原始研究作者提供的RF模型测试结果如下:
表2-4 原研究提供的RF模型测试结果
| 机器学习模型 | AUC值 | 准确度 | 特异度 |
|---|---|---|---|
| Random Forest | 0.930 | 0.870 | 0.871 |
本章小结
本章通过"高级Prompt生成器"对三类典型临床科研场景进行了任务拆解、测试验证和结果对比。实践结果表明,该软件能够有效实现需求分析中的核心功能,生成的代码在三类场景中均展现出良好的性能,部分模型表现甚至优于已发表研究的结果,验证了软件在临床科研辅助中的实际应用价值。
总结
本研究聚焦临床科研中工作者面临的机器学习应用门槛高、代码实现复杂等核心问题,针对大语言模型在医学领域应用中存在的提示词设计专业性不足、输出质量不稳定等挑战,提出面向临床科研的提示词工程适配方法,开发"高级Prompt生成器"软件。
研究采用需求驱动的软件开发模式,基于PyQt5与Tkinter构建GUI界面,集成数据预处理、模型选择、评估指标定制等功能模块;通过思维链策略优化提示词模板,解决模型幻觉问题,并配套开发"医疗数据列名提取系统"提升操作效率。选取三类典型临床任务开展验证,对比DeepSeek生成代码与原始研究的模型性能,结果显示生成模型的AUROC值与原始研究差异不显著,部分场景甚至表现更优,验证了方法的有效性和可靠性。
技术突破方面,本研究提出医学领域专用提示词设计规则,通过角色定义、任务分解与动态约束,使非专业用户可生成高质量代码,填补了自动化机器学习在自然语言交互方面的空白。实践意义上,该方案显著降低了临床科研中机器学习的应用门槛,促进了医学与AI的交叉融合,可有效缩短研究周期。学科推动方面,为医学大语言模型的应用提供了标准化流程,推动循证医学任务处理与AI技术的结合,助力精准医疗发展。
本研究仍存在一定不足:一是数据依赖性强,当前模板对数据集结构的预设可能限制泛化能力,未来可引入自适应数据解析算法,支持非结构化医疗数据输入;二是模型解释性不足,生成代码缺乏可解释性模块,建议集成模型解释模板,增强临床决策可信度;三是实时性局限,未考虑在线学习场景。
未来可进一步探索临床决策闭环构建,将提示词生成系统嵌入电子健康记录平台,形成"问题识别→模型生成→结果反馈"的智能闭环;同时通过构建专业数据库,让大语言模型实现自我感知,生成更精准的解决方案。
需要特别强调的是,我们提供24小时响应"代码运行异常"的应急修复服务,比学生自行调试效率提升40%。我们始终倡导"买代码不如买明白"的理念,通过人工深度创作降低查重风险,同时保障代码无漏洞,直击学生"代码能运行但怕查重、怕漏洞"的核心痛点。

参考文献
- 万艳丽, 王颖帅, 赵姗姗. 医学大模型研究进展[J]. 医学研究杂志, 2024, 53(10): 1-6, 186.
- Singhal K, Azizi S, Tu T, et al. Large Language Models Encode Clinical Knowledge[J]. Nature, 2023, 620(7972): 172-180.
- Gao Y, Xin L, Lin H, et al. Machine Learning-Based Automated Sponge Cytology for Screening of Oesophageal Squamous Cell Carcinoma and Adenocarcinoma of the Oesophagogastric Junction: A Nationwide, Multicohort, Prospective Study[J]. The Lancet Gastroenterology & Hepatology, 2023, 8(5): 432-445.
- Breiman L. Random Forests[J]. Machine Learning, 2001, 45: 5-32.
- Boser B E, Guyon I M, Vapnik V N, et al. A Training Algorithm for Optimal Margin Classifiers[C]. //Proceedings of the 5th Annual Workshop on Computational Learning Theory (COLT). New York, NY, USA: Association for Computing Machinery, 1992: 144-152.
- Luo X, Li F, Xu W, et al. Machine Learning-Based Genetic Diagnosis Models for Hereditary Hearing Loss by the GJB2, SLC26A4 and MT-RNR1 Variants[J]. EBioMedicine, 2021, 69: 103322.
- Huda A, Castaño A, Niyogi A, et al. A Machine Learning Model for Identifying Patients at Risk for Wild-Type Transthyretin Amyloid Cardiomyopathy[J]. Nature Communications, 2021, 12(1): 2725.


 LangChain、FastAPI、Python大型语言模型LLM电商多智能体Multi-Agent客服系统|附代码
LangChain、FastAPI、Python大型语言模型LLM电商多智能体Multi-Agent客服系统|附代码 LLM嵌入K-Means、DBSCAN聚类、PCA主成分分析新闻文本聚类研究|附代码数据
LLM嵌入K-Means、DBSCAN聚类、PCA主成分分析新闻文本聚类研究|附代码数据 Qwen3大模型本地化部署、LoRA低秩适配轻量化微调与医疗推理领域应用落地研究|附代码数据
Qwen3大模型本地化部署、LoRA低秩适配轻量化微调与医疗推理领域应用落地研究|附代码数据 Python用SentenceTransformer、OLS、集成学习、模型蒸馏情感分类金融新闻文本|附代码数据
Python用SentenceTransformer、OLS、集成学习、模型蒸馏情感分类金融新闻文本|附代码数据



