自组织映射神经网络(SOM)是一种无监督的数据可视化技术,可用于可视化低维(通常为2维)表示形式的高维数据集。
自组织映射神经网络(Self-Organizing Map,简称SOM),也称为Kohonen网络,是一种无监督学习算法,用于数据的降维和可视化。SOM通过将高维数据映射到低维空间(通常是二维的网格),来保持数据的拓扑结构,即相似的输入数据点会被映射到网格上的相邻位置。
SOM网络由输入层和竞争层(或映射层)组成。输入层负责接收原始的高维数据,而竞争层则是由一组神经元构成的二维网格,这些神经元会学习如何最好地表示输入数据。
可下载资源
作者

SOM
SOM可视化由多个“节点”组成。每个节点向量具有:
- 在SOM网格上的位置
- 与输入空间维度相同的权重向量。(例如,如果您的输入数据代表人,则可能具有变量“年龄”,“性别”,“身高”和“体重”,网格上的每个节点也将具有这些变量的值)
- 输入数据中的关联样本。输入空间中的每个样本都“映射”或“链接”到网格上的节点。一个节点可以代表多个输入样本。
SOM的关键特征是原始输入数据的拓扑特征保留在图上。这意味着将相似的输入样本(其中相似性是根据输入变量(年龄,性别,身高,体重)定义的)一起放置在SOM网格上。
例如,所有高度大约为1.6m的55岁女性将被映射到网格同一区域中的节点。考虑到所有变量,身材矮小的人将被映射到其他地方。在身材上,高个的男性比小个的胖男性更接近高个头的女性,因为他们“相似”得多。
SOM热图
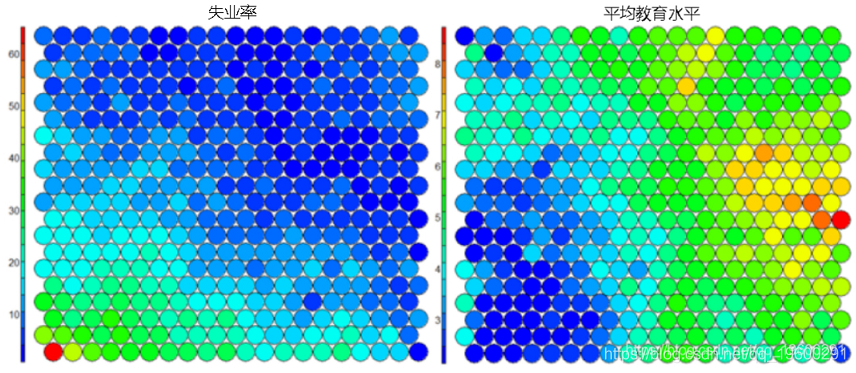
典型的SOM可视化是“热图”。热图显示了变量在SOM中的分布。理想情况下,相似年龄的人应该聚集在同一地区。
下图使用两个热图说明平均教育水平和失业率之间的关系。
SOM算法
从样本数据集生成SOM的算法可总结如下:
- 选择地图的大小和类型。形状可以是六边形或正方形,具体取决于所需节点的形状。通常,最好使用六边形网格,因为每个节点都具有6个近邻 。
- 随机初始化所有节点权重向量。
- 从训练数据中选择一个随机数据点,并将其呈现给SOM。
- 在地图上找到“最佳匹配单位”(BMU)–最相似的节点。使用欧几里德距离公式计算相似度。
- 确定BMU“邻居”内的节点。
–邻域的大小随每次迭代而减小。 - 所选数据点调整BMU邻域中节点的权重。
–学习率随着每次迭代而降低。
–调整幅度与节点与BMU的接近程度成正比。 - 重复步骤2-5,进行N次迭代/收敛。
R中的SOM
训练
R可以创建SOM和可视化。
# 在R中创建自组织映射
# 创建训练数据集(行是样本,列是变量
# 在这里,我选择“数据”中可用的变量子集
data_train <- data[, c(3,4,5,8)]
#将带有训练数据的数据框更改为矩阵
#同时对所有变量进行标准化
#SOM训练过程。
data_train_matrix <- as.matrix(scale(data_train))
#创建SOM网格
#在训练SOM之前先训练网格
grid(xdim = 20, ydim=20, topo="hexagonal")
#最后,训练SOM,迭代次数选项,
#学习率
model <- som(data_train_matrix)可视化
可视化可以检察生成SOM的质量,并探索数据集中变量之间的关系。
训练过程
随着SOM训练迭代的进行,从每个节点的权重到该节点表示的样本的距离将减小。理想情况下,该距离应达到最小。此图选项显示了随着时间的进度。如果曲线不断减小,则需要更多的迭代。
#SOM的训练进度
plot(model, type="changes")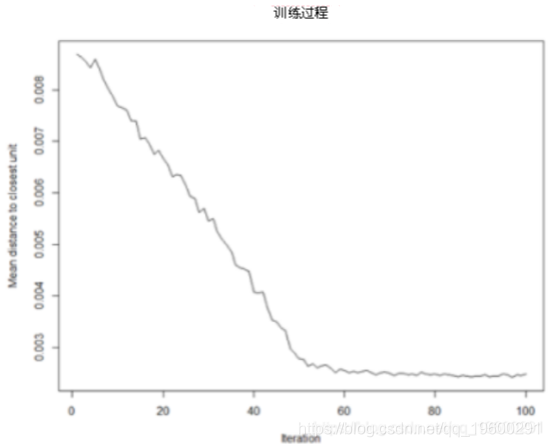
节点计数
我们可以可视化映射到地图上每个节点的样本数。此度量可以用作图质量的度量-理想情况下,样本分布相对均匀。选择图大小时,每个节点至少要有5-10个样本。
#节点数
plot(model, type="count")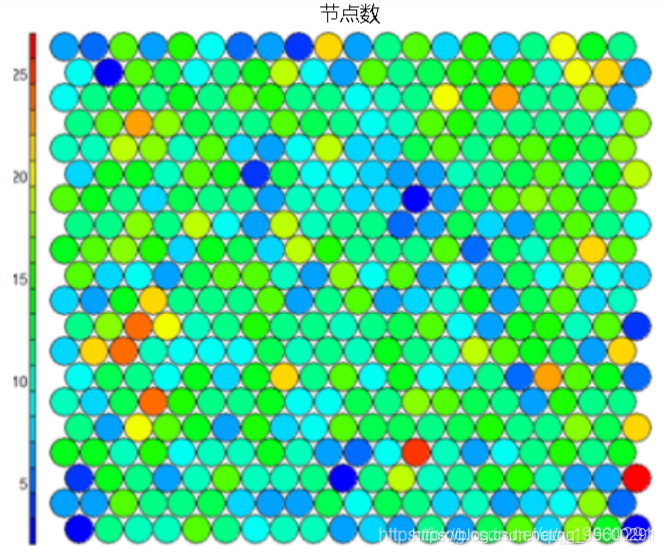
邻居距离
通常称为“ U矩阵”,此可视化表示每个节点与其邻居之间的距离。通常使用灰度查看,邻居距离低的区域表示相似的节点组。距离较大的区域表示节点相异得多。U矩阵可用于识别SOM映射内的类别。
# U-matrix 可视化

代码/权重向量
节点权重向量由用于生成SOM的原始变量值。每个节点的权重向量代表/相似于映射到该节点的样本。通过可视化整个地图上的权重向量,我们可以看到样本和变量分布中的模型。权重向量的默认可视化是一个“扇形图”,其中为每个节点显示了权重向量中每个变量的大小的各个扇形表示。
# 权重矢量视图

热图
热图是也许是自组织图中最重要的可能的可视化。通常,SOM过程创建多个热图,然后比较这些热图以识别图上有趣的区域。
在这种情况下,我们将SOM的平均教育水平可视化。
# 热图创建
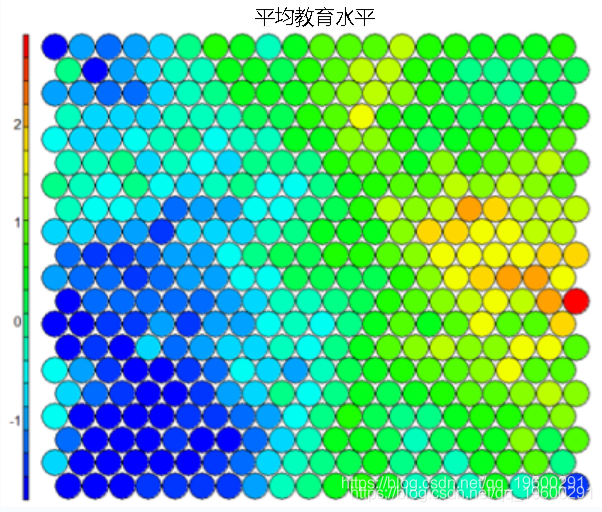
应该注意的是,该默认可视化绘制了感兴趣变量的标准化版本。
# 未标准化的热图
#定义要绘制的变量
aggregate(as.numeric(data_train, by=list(som_model$unit.classi FUN=mean)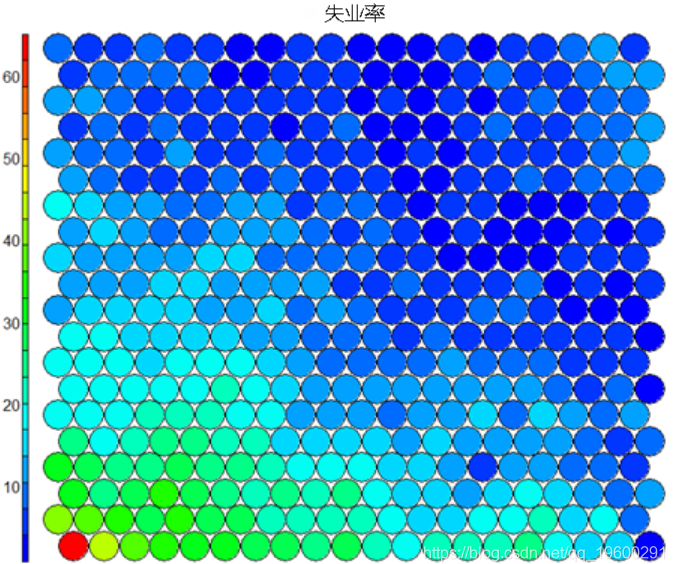
值得注意的是,上面的热图显示了失业率与教育水平之间的反比关系。并排显示的其他热图可用于构建不同区域及其特征的图片。
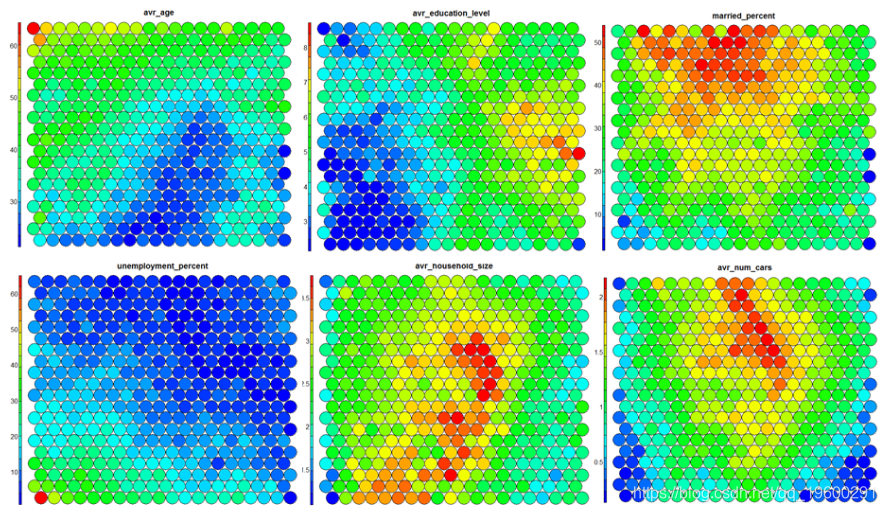
SOM网格中具有空节点的热图
在某些情况下,您的SOM训练可能会导致SOM图中的节点为空。通过几行,我们可以找到som_model $ unit.classif中缺少的节点,并将其替换为NA值–此步骤将防止空节点扭曲您的热图。
# 当SOM中有空节点时绘制未标准化的变量
var_unscaled <- aggregate(as.numeric(data_train_raw), by=list(som_model$unit.classif), FUN=mean)
# 为未分配的节点添加NA值
missingNodes <- which(!(seq(1,nrow(som_model$codes) %in% varunscaled$Node))
# 将它们添加到未标准化的数据框
var_unscaled <- rbind(var_unscaled, data.frame(Node=missingNodes, Value=NA))
# 结果数据框
var_unscaled
# 现在仅使用正确的“值”创建热图。
plot(som_model, type =d)自组织图的聚类和分割
可以在SOM节点上执行聚类,以发现具有相似度量的样本组。可以使用kmeans算法并检查“类内平方和之内”图中的“肘点”来确定合适的聚类数估计。
# 查看WCSS的kmeans
for (i in 2:15) {
wss[i] <- sum(kmeans(mydata, centers=i)$withinss)
}
# 可视化聚类结果
##使用分层聚类对向量进行聚类
cutree(hclust(dist(som_model$codes)), 6)
# 绘制这些结果:
plot(som_model, t"mappinol =ty_pal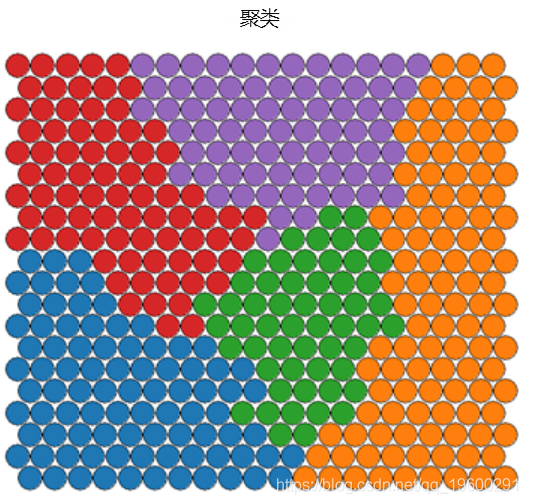
理想情况下,发现的类别在图表面上是连续的。为了获得连续的聚类,可以使用仅在SOM网格上仅将相似AND的节点组合在一起的层次聚类算法。
将聚类映射回原始样本
当按照上面的代码示例应用聚类算法时,会将聚类分配给 SOM映射上的每个 节点,而不是 数据集中的原始 样本。
# 为每个原始数据样本获取具有聚类值的向量
som_clust[som_modl$unit.clasf]
# 为每个原始数据样本获取具有聚类值的向量
data$cluster <- cluster_assignment使用每个聚类中训练变量的统计信息和分布来构建聚类特征的有意义的图片-这既是艺术又是科学,聚类和可视化过程通常是一个迭代过程。
结论
自组织映射(SOM)是数据科学中的一个强大工具。优势包括:
- 发现客户细分资料的直观方法。
- 相对简单的算法,易于向非数据科学家解释结果
- 可以将新的数据点映射到经过训练的模型以进行预测。
缺点包括:
- 由于训练数据集是迭代的,因此对于非常大的数据集缺乏并行化功能
- 很难在二维平面上表示很多变量
- SOM训练需要清理后的,数值的数据,这些数据很难获得。



 R语言层次聚类、多维缩放MDS分类RNA测序(RNA-seq)乳腺发育基因数据可视化|附数据代码
R语言层次聚类、多维缩放MDS分类RNA测序(RNA-seq)乳腺发育基因数据可视化|附数据代码 SPSS大学生网络购物行为研究:因子分析、主成分、聚类、交叉表和卡方检验
SPSS大学生网络购物行为研究:因子分析、主成分、聚类、交叉表和卡方检验 R语言聚类分析、因子分析、主成分分析PCA农村农业相关经济指标数据可视化
R语言聚类分析、因子分析、主成分分析PCA农村农业相关经济指标数据可视化 R语言拟合线性混合效应模型、固定效应、随机效应参数估计可视化生物生长、发育、繁殖影响因素
R语言拟合线性混合效应模型、固定效应、随机效应参数估计可视化生物生长、发育、繁殖影响因素


