
最近我们被客户要求撰写关于层次聚类的研究报告。
本文在iris数据集上展示了如何使用R软件来建立Hierarchical Cluster Analysis层次聚类分析(更好的可视化和灵敏度分析)。
可下载资源
背景
鸢尾花数据集
该鸢尾花数据集是学习监督分类算法的常用数据。
通过以下散点图矩阵(SPLOM)可以轻松看出这一点:
#加载数据
iris <- datasets::iris
iris2 <- iris[,-5]
species_labels <- iris[,5]
library(colorspace) # 颜色包
species_col <- rev(rainbow_hcl(3))[as.numeric(species_labels)]
#绘制 SPLOM:
pairs(iris2, col = species_col,
lower.panel = NULL,
cex.labels=2, pch=19, cex = 1.2)
# 添加图例
par(xpd = TRUE)
legend(x = 0.05, y = 0.4, cex = 2,
legend = as.character(levels(species_labels)),
fill = unique(species_col))
par(xpd = NA)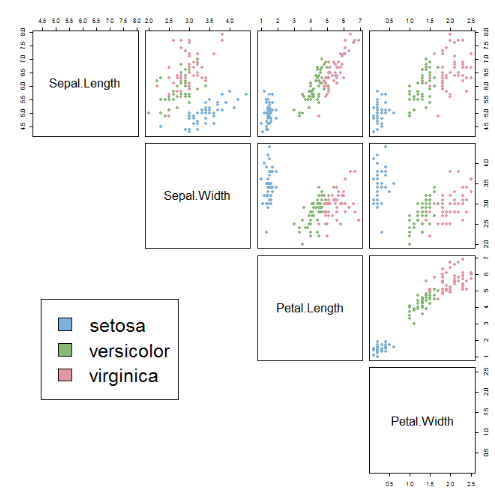
我们可以看到,Setosa物种与Versicolor和Virginica明显不同(它们具有较低的花瓣长度和宽度)。但是,基于对萼片和花瓣宽度/长度的测量,不易将Versicolor和Virginica分开。
通过查看数据的平行坐标图可以得出同样的结论:
par(las = 1, mar = c(4.5, 3, 3, 2) + 0.1, cex = .8)
MASS::parcoord(iris2, col = species_col, var.label = TRUE, lwd = 2)
# 添加标题
title("Parallel coordinates plot of the Iris data")
# 添加图例
par(xpd = TRUE)
legend(x = 1.75, y = -.25, cex = 1,
legend = as.character(levels(species_labels)),
fill = unique(species_col), horiz = TRUE)
我们可以通过将对象转化为树状图并对对象进行一些调整来可视化运行它的结果
相同的可以在圆形布局中呈现:
这些可视化很容易证明分层聚类的分离对于“Setosa”物种来说是非常好的,但是在将许多“Versicolor”物种标记为“Virginica”时未能实现。
我们也可以使用热图探索数据。
在热图中,我们还可以看到Setosa物种的花瓣值如何(浅黄色),但很难看出其他两种物种之间的明显区别。
各种聚类算法之间的相似/不同
为了进行这种分析,我们将创建所有8个hclust对象,并将它们链接在一起成为一个dendlist对象(顾名思义,它可以将一组树状图组合在一起用于进一步分析)。
接下来,我们可以看看每个聚类结果之间的同源相关性cor.dendlist。(这可以使用corrplot包中的corrplot函数很好地绘制):
从上图中我们可以很容易地看到,除了完整的方法(默认方法in hclust),大多数聚类方法的结果非常相似,其结果相关度大约为0.6。
随时关注您喜欢的主题
默认的同源相关使用皮尔逊的度量,但如果我们使用spearman的相关系数呢?
我们可以看到,相关性不是很强,表明一个行为依赖于彼此距离很远的一些项目,这些项目对皮尔森相关性的影响相关性更大。


 R语言层次聚类、多维缩放MDS分类RNA测序(RNA-seq)乳腺发育基因数据可视化|附数据代码
R语言层次聚类、多维缩放MDS分类RNA测序(RNA-seq)乳腺发育基因数据可视化|附数据代码 SPSS大学生网络购物行为研究:因子分析、主成分、聚类、交叉表和卡方检验
SPSS大学生网络购物行为研究:因子分析、主成分、聚类、交叉表和卡方检验 R语言聚类分析、因子分析、主成分分析PCA农村农业相关经济指标数据可视化
R语言聚类分析、因子分析、主成分分析PCA农村农业相关经济指标数据可视化 R语言、WEKA关联规则、决策树、聚类、回归分析工业企业创新情况影响因素数据
R语言、WEKA关联规则、决策树、聚类、回归分析工业企业创新情况影响因素数据


